46 整洁建模基础
从前面的模型介绍可以看出, 常见的有监督学习问题为回归问题和判别问题, 每一种问题都可以用许多种不同模型解决, 而每一个模型都可能包含需要调优的超参数集合, 对数据可能有不同的预处理、转码、函数变换、降维等变换。 这些模型由许多个R扩展包实现, 而且接口、选项各自不同。
tidymodels包提供了针对统计建模用户简便易用的建模功能和使用规范。
R有许多建模用的扩展包,
不同的扩展包在解决类似的回归、判别问题时,
可能用了相近但不相同,
或者完全不同的界面接口。
tidymodels试图统一这些接口,
使得用户可以用相同的方法调用不同的计算包。
tidyr包的broom函数、nest、unnest函数提供了数据框分组建模的方便功能。
参见
- Max Kuhn and Julia Silge: Tidy Modeling with R https://www.tmwr.org/
46.1 建模一般步骤
在建模之前, 需要先获取数据。 统计建模用的数据一般都保存为数据框格式。
必须对数据有充分的了解, 否则在后续使用模型时, 就无法判断模型所对应的各种理论假定是否满足, 比如, 数据是否能够认为是独立的, 数据是否没有偏差地代表了所要研究的对象。
获得数据后, 一般需要花费时间进行“数据清理”。 数据中可能有许多错误、不一致、缺失内容, 都需要进行整理修改。 对缺失数据, 常见的办法是仅保留没有任何缺失内容的案例, 但是这常常会使得保留的数据量太小, 另外有可能删去的内容使得保留的数据带有偏差。 另一种对待缺失数据的方法是进行插补, 这又有简单插补, 如用均值代替, 与利用相关性插补等方法。
在建模之前, 应增加对数据的直观了解, 比如, 数据的收集方式, 每个属性(变量)的可取值范围, 变量分布情况, 变量之间的关系, 观测有没有明显分组聚集, 等等。 这些研究称为探索性数据分析(EDA)。
随后的工作是建模、评估、使用。 模型大致可以分为:
- 描述性模型, 如描述统计量,直方图,散点图,散点图局部曲线拟合。
- 推断性模型,如置信区间、假设检验、贝叶斯决策分析等。 推断性模型的结论一般是带有概率判断的。
- 预测性模型,如回归分析、判别分析等。主要关心预测的精度。 有些模型基于数学方程,如微分方程、回归模型, 有些模型所需的模型假设很少, 如最近邻回归。
建模的时候, 一般不仅仅考虑一个模型, 而是比较多个模型; 对同一个模型, 也有一些模型参数需要进行调整使得模型性能最优, 比如lasso回归中的光滑性系数的选择, 提升法(boosting)中学习速度和停止时间的选择。 模型比较和参数调优经常使用将数据交叉划分来评估性能的方法。
在研究模型表现时, 有可能会发现一些数据中的不足, 这时有可能需要对数据进行一些新的变换, 或进一步地去收集更多的数据。
在比较模型并筛选出少数较优模型后, 应对模型的性能进行更客观的判断, 如果是预报问题, 这通常需要使用建模时未使用过的数据来评估模型的表现。
46.1.1 训练集和测试集的划分
统计建模(统计学习)问题, 一般都需要将所有数据划分为两个子集:
- 训练集(training set)。 所有的探索性分析、模型试验、参数调整、模型比较都在训练集上进行, 从训练集产生一两个最成功的模型。
- 测试集(testing set)。 仅在训练完成后才将模型应用在测试集上, 评估模型的性能。 训练阶段不允许访问测试集, 一旦因为某种目的在训练阶段使用了测试集, 测试集就失效了。
划分训练集和测试集, 常用如下三种方法:
- 从\(n\)个观测中用随机无放回抽样抽取指定比例的训练集,
余集为测试集。
可以使用基本R函数
sample()获取训练集的下标集合。 - 对于因变量为分类变量且类别比例相差悬殊的情形, 可以按因变量类别进行分层抽样,组合成训练集, 然后余集为测试集。 这也适用于因变量为连续取值但分布重尾的情形。
- 对于沿时间记录的数据, 只能以前面一段时间为训练集, 后面剩余的时间为测试集, 不能选择中间的时间为测试集。
R扩展包rsample提供了对数据框划分训练集和测试集的函数initial_split。
为了用简单随机抽样划分,
用法如:
其中prop指定训练集比例,
da是原始数据框。
为了提取训练集和测试集,
用法如
为了按因变量y的不同类别进行分层抽样产生训练集,
用法如:
这也适用于y是连续取值的情形,
将自动按y的四分位数将y取值范围分成4份进行分层抽样。
对于数据框的各行是按时间顺序记录的情形, 划分训练集和测试集可用如:
有些数据, 如纵向数据(longitudinal data), 每个受试者(观测单元)有多次观测, 这些观测不独立, 需要以受试者为单位进行随机抽取, 而不是以观测为单位进行抽取。
46.2 用parsnip建模
不同的R建模扩展包使用不同的访问接口, parsnip包则可以将这些扩展包作为计算引擎, 给用户提供一个统一的访问界面。
46.2.1 Ames数据例子
用modeldata扩展包的ames数据集为例。
## Rows: 2,930
## Columns: 74
## $ MS_SubClass <fct> One_Story_1946_and_Newer_All_Styles, One_Story_1946…
## $ MS_Zoning <fct> Residential_Low_Density, Residential_High_Density, …
## $ Lot_Frontage <dbl> 141, 80, 81, 93, 74, 78, 41, 43, 39, 60, 75, 0, 63,…
## $ Lot_Area <int> 31770, 11622, 14267, 11160, 13830, 9978, 4920, 5005…
## $ Street <fct> Pave, Pave, Pave, Pave, Pave, Pave, Pave, Pave, Pav…
## $ Alley <fct> No_Alley_Access, No_Alley_Access, No_Alley_Access, …
## $ Lot_Shape <fct> Slightly_Irregular, Regular, Slightly_Irregular, Re…
## $ Land_Contour <fct> Lvl, Lvl, Lvl, Lvl, Lvl, Lvl, Lvl, HLS, Lvl, Lvl, L…
## $ Utilities <fct> AllPub, AllPub, AllPub, AllPub, AllPub, AllPub, All…
## $ Lot_Config <fct> Corner, Inside, Corner, Corner, Inside, Inside, Ins…
## $ Land_Slope <fct> Gtl, Gtl, Gtl, Gtl, Gtl, Gtl, Gtl, Gtl, Gtl, Gtl, G…
## $ Neighborhood <fct> North_Ames, North_Ames, North_Ames, North_Ames, Gil…
## $ Condition_1 <fct> Norm, Feedr, Norm, Norm, Norm, Norm, Norm, Norm, No…
## $ Condition_2 <fct> Norm, Norm, Norm, Norm, Norm, Norm, Norm, Norm, Nor…
## $ Bldg_Type <fct> OneFam, OneFam, OneFam, OneFam, OneFam, OneFam, Twn…
## $ House_Style <fct> One_Story, One_Story, One_Story, One_Story, Two_Sto…
## $ Overall_Cond <fct> Average, Above_Average, Above_Average, Average, Ave…
## $ Year_Built <int> 1960, 1961, 1958, 1968, 1997, 1998, 2001, 1992, 199…
## $ Year_Remod_Add <int> 1960, 1961, 1958, 1968, 1998, 1998, 2001, 1992, 199…
## $ Roof_Style <fct> Hip, Gable, Hip, Hip, Gable, Gable, Gable, Gable, G…
## $ Roof_Matl <fct> CompShg, CompShg, CompShg, CompShg, CompShg, CompSh…
## $ Exterior_1st <fct> BrkFace, VinylSd, Wd Sdng, BrkFace, VinylSd, VinylS…
## $ Exterior_2nd <fct> Plywood, VinylSd, Wd Sdng, BrkFace, VinylSd, VinylS…
## $ Mas_Vnr_Type <fct> Stone, None, BrkFace, None, None, BrkFace, None, No…
## $ Mas_Vnr_Area <dbl> 112, 0, 108, 0, 0, 20, 0, 0, 0, 0, 0, 0, 0, 0, 0, 6…
## $ Exter_Cond <fct> Typical, Typical, Typical, Typical, Typical, Typica…
## $ Foundation <fct> CBlock, CBlock, CBlock, CBlock, PConc, PConc, PConc…
## $ Bsmt_Cond <fct> Good, Typical, Typical, Typical, Typical, Typical, …
## $ Bsmt_Exposure <fct> Gd, No, No, No, No, No, Mn, No, No, No, No, No, No,…
## $ BsmtFin_Type_1 <fct> BLQ, Rec, ALQ, ALQ, GLQ, GLQ, GLQ, ALQ, GLQ, Unf, U…
## $ BsmtFin_SF_1 <dbl> 2, 6, 1, 1, 3, 3, 3, 1, 3, 7, 7, 1, 7, 3, 3, 1, 3, …
## $ BsmtFin_Type_2 <fct> Unf, LwQ, Unf, Unf, Unf, Unf, Unf, Unf, Unf, Unf, U…
## $ BsmtFin_SF_2 <dbl> 0, 144, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 1120, 0…
## $ Bsmt_Unf_SF <dbl> 441, 270, 406, 1045, 137, 324, 722, 1017, 415, 994,…
## $ Total_Bsmt_SF <dbl> 1080, 882, 1329, 2110, 928, 926, 1338, 1280, 1595, …
## $ Heating <fct> GasA, GasA, GasA, GasA, GasA, GasA, GasA, GasA, Gas…
## $ Heating_QC <fct> Fair, Typical, Typical, Excellent, Good, Excellent,…
## $ Central_Air <fct> Y, Y, Y, Y, Y, Y, Y, Y, Y, Y, Y, Y, Y, Y, Y, Y, Y, …
## $ Electrical <fct> SBrkr, SBrkr, SBrkr, SBrkr, SBrkr, SBrkr, SBrkr, SB…
## $ First_Flr_SF <int> 1656, 896, 1329, 2110, 928, 926, 1338, 1280, 1616, …
## $ Second_Flr_SF <int> 0, 0, 0, 0, 701, 678, 0, 0, 0, 776, 892, 0, 676, 0,…
## $ Gr_Liv_Area <int> 1656, 896, 1329, 2110, 1629, 1604, 1338, 1280, 1616…
## $ Bsmt_Full_Bath <dbl> 1, 0, 0, 1, 0, 0, 1, 0, 1, 0, 0, 1, 0, 1, 1, 1, 0, …
## $ Bsmt_Half_Bath <dbl> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
## $ Full_Bath <int> 1, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 1, 1, 3, 2, …
## $ Half_Bath <int> 0, 0, 1, 1, 1, 1, 0, 0, 0, 1, 1, 0, 1, 1, 1, 1, 0, …
## $ Bedroom_AbvGr <int> 3, 2, 3, 3, 3, 3, 2, 2, 2, 3, 3, 3, 3, 2, 1, 4, 4, …
## $ Kitchen_AbvGr <int> 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, 1, …
## $ TotRms_AbvGrd <int> 7, 5, 6, 8, 6, 7, 6, 5, 5, 7, 7, 6, 7, 5, 4, 12, 8,…
## $ Functional <fct> Typ, Typ, Typ, Typ, Typ, Typ, Typ, Typ, Typ, Typ, T…
## $ Fireplaces <int> 2, 0, 0, 2, 1, 1, 0, 0, 1, 1, 1, 0, 1, 1, 0, 1, 0, …
## $ Garage_Type <fct> Attchd, Attchd, Attchd, Attchd, Attchd, Attchd, Att…
## $ Garage_Finish <fct> Fin, Unf, Unf, Fin, Fin, Fin, Fin, RFn, RFn, Fin, F…
## $ Garage_Cars <dbl> 2, 1, 1, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 2, 3, 2, …
## $ Garage_Area <dbl> 528, 730, 312, 522, 482, 470, 582, 506, 608, 442, 4…
## $ Garage_Cond <fct> Typical, Typical, Typical, Typical, Typical, Typica…
## $ Paved_Drive <fct> Partial_Pavement, Paved, Paved, Paved, Paved, Paved…
## $ Wood_Deck_SF <int> 210, 140, 393, 0, 212, 360, 0, 0, 237, 140, 157, 48…
## $ Open_Porch_SF <int> 62, 0, 36, 0, 34, 36, 0, 82, 152, 60, 84, 21, 75, 0…
## $ Enclosed_Porch <int> 0, 0, 0, 0, 0, 0, 170, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0…
## $ Three_season_porch <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
## $ Screen_Porch <int> 0, 120, 0, 0, 0, 0, 0, 144, 0, 0, 0, 0, 0, 0, 140, …
## $ Pool_Area <int> 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, 0, …
## $ Pool_QC <fct> No_Pool, No_Pool, No_Pool, No_Pool, No_Pool, No_Poo…
## $ Fence <fct> No_Fence, Minimum_Privacy, No_Fence, No_Fence, Mini…
## $ Misc_Feature <fct> None, None, Gar2, None, None, None, None, None, Non…
## $ Misc_Val <int> 0, 0, 12500, 0, 0, 0, 0, 0, 0, 0, 0, 500, 0, 0, 0, …
## $ Mo_Sold <int> 5, 6, 6, 4, 3, 6, 4, 1, 3, 6, 4, 3, 5, 2, 6, 6, 6, …
## $ Year_Sold <int> 2010, 2010, 2010, 2010, 2010, 2010, 2010, 2010, 201…
## $ Sale_Type <fct> WD , WD , WD , WD , WD , WD , WD , WD , WD , WD , W…
## $ Sale_Condition <fct> Normal, Normal, Normal, Normal, Normal, Normal, Nor…
## $ Sale_Price <int> 215000, 105000, 172000, 244000, 189900, 195500, 213…
## $ Longitude <dbl> -93.61975, -93.61976, -93.61939, -93.61732, -93.638…
## $ Latitude <dbl> 42.05403, 42.05301, 42.05266, 42.05125, 42.06090, 4…后面将因变量变成房屋售价的常用对数值。
划分训练集和测试集:
set.seed(101)
ames_split <- initial_split(ames, prop = 0.80)
ames_train <- training(ames_split)
ames_test <- testing(ames_split)
dim(ames_train)## [1] 2344 7446.2.2 线性回归示例
parsnip包用类似的函数名和语法指定要使用的模型与计算引擎。 这会指定如下的选择:
- 模型的数学背景,如线性回归、随机森林、KNN等;
- 模型的计算引擎,这一般是规定要使用的R扩展包;
- 模型是回归还判别问题。 如果某个计算引擎只能处理其中一种问题, 则指定了计算引擎也选择了要处理的问题。
这些选择并不与数据集和变量绑定。
例如,
选择最小二乘的线性回归模型,
使用基本R的lm()函数作为计算引擎:
其中的tidymodels_prefer()用于载入的多个扩展包的名字冲突的解决,
可以用tidymodels默认的一些冲突解决方案。
下面将数据、变量与模型联系在一起, 在训练集上产生模型拟合结果:
## parsnip model object
##
##
## Call:
## stats::lm(formula = Sale_Price ~ Longitude + Latitude, data = data)
##
## Coefficients:
## (Intercept) Longitude Latitude
## -301.337 -1.986 2.869fit()函数的一个变种是fit_xy(),
适用于自变量保存为矩阵或数据框,
因变量保存为向量的情形,如:
lm_xy_fit <-
lm_model |>
fit_xy(
x = ames_train |> select(Longitude, Latitude),
y = ames_train |> pull(Sale_Price)
)
lm_xy_fit## parsnip model object
##
##
## Call:
## stats::lm(formula = ..y ~ ., data = data)
##
## Coefficients:
## (Intercept) Longitude Latitude
## -301.337 -1.986 2.869fit()和fit_xy()的区别是,
fit_xy()不对输入数据进行变换,
直接传递给计算引擎,
而fit()在计算引擎要求输入矩阵时,
会对输入数据进行变换,
比如将类别变量转换为哑变量。
46.2.3 随机森林示例
parsnip对于同一模型的不同计算引擎提供了模型参数的统一设置格式。
比如,
随机森林算法,
需要设置每次随机选择的自变量个数、合并的树的个数、每棵树的大小,
不同的随机森林扩展包如ranger, randomForest, sparklyr对这些设置使用不同的参数名称。
parsnip的rand_forest()函数则可以用统一的格式指定这些参数,
允许使用不同的引擎。
如:
这里min_n代表每个叶结点包含观测个数的下限,
低于此下限不能继续拆分,
trees代表树的数目,
mtry代表每次分叉可以从多少个自变量中随机选择。
可以用translate()函数查看parsnip的参数设置如何映射到计算引擎包本来的设置,
如:
## Random Forest Model Specification (regression)
##
## Main Arguments:
## trees = 1000
## min_n = 5
##
## Computational engine: ranger
##
## Model fit template:
## ranger::ranger(x = missing_arg(), y = missing_arg(), weights = missing_arg(),
## num.trees = 1000, min.node.size = min_rows(~5, x), num.threads = 1,
## verbose = FALSE, seed = sample.int(10^5, 1))可见在ranger包中trees变成了num.trees,
min_n变成了min.node.size。
有一些参数是各个引擎特有的,
这在parsnip中会自动设置一些合适的值,
也可以在set_engine()函数中添加,
如set_engine("ranger", verbose = TRUE)。
46.2.4 提取拟合结果
extract_fit_engine()从fit()和fit_xy()的结果中提取所使用引擎输出的拟合结果,
然后可以利用基本R本身的模型信息提取函数如coef(), vcov()等提取结果内容,如:
## (Intercept) Longitude Latitude
## -301.337454 -1.986050 2.868594## (Intercept) Longitude Latitude
## (Intercept) 203.734986 1.5807903348 -1.3252325337
## Longitude 1.580790 0.0168109489 -0.0001562159
## Latitude -1.325233 -0.0001562159 0.0311793054##
## Call:
## stats::lm(formula = Sale_Price ~ Longitude + Latitude, data = data)
##
## Residuals:
## Min 1Q Median 3Q Max
## -1.02459 -0.09740 -0.01793 0.10037 0.57726
##
## Coefficients:
## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -301.3375 14.2736 -21.11 <2e-16 ***
## Longitude -1.9861 0.1297 -15.32 <2e-16 ***
## Latitude 2.8686 0.1766 16.25 <2e-16 ***
## ---
## Signif. codes: 0 '***' 0.001 '**' 0.01 '*' 0.05 '.' 0.1 ' ' 1
##
## Residual standard error: 0.1597 on 2341 degrees of freedom
## Multiple R-squared: 0.1746, Adjusted R-squared: 0.1739
## F-statistic: 247.6 on 2 and 2341 DF, p-value: < 2.2e-16## Estimate Std. Error t value Pr(>|t|)
## (Intercept) -301.337454 14.2735765 -21.11156 1.049258e-90
## Longitude -1.986050 0.1296570 -15.31772 1.514774e-50
## Latitude 2.868594 0.1765766 16.24561 2.569212e-56这些信息提取函数有各自的用处, 缺点是返回值类型很不统一, 而且不同引擎对信息提取函数的支持也不相同。 broom包的tidy函数可以将模型拟合结果统一转化为数据框形式, 方便了后续的利用。 如:
## # A tibble: 3 × 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) -301. 14.3 -21.1 1.05e-90
## 2 Longitude -1.99 0.130 -15.3 1.51e-50
## 3 Latitude 2.87 0.177 16.2 2.57e-5646.2.5 预测
parsnip实现的predict()函数,
总是以模型拟合结果为输入,
用new_data输入要预测的数据,
输出结果十分规范:
- 结果总是一个tibble数据框;
- 结果的行数总是等于输入数据长度, 如果输入数据有缺失值, 则对应结果取缺失值;
- 数据框变量名总是有明显含义的。
这大大减轻了用户的学习成本, 因为许多机器学习扩展包提供的预测结果是非标准的, 需要用户自己想办法提取需要的预测内容。
输出结果数据框包含的变量规定如下:
- 若因变量为数值型,
要做点预测,
选
type = "numeric", 结果变量为.pred; - 因变量为类别,
要预测类别,
选
type = "class", 结果变量为.pred_class; - 若因变量为类别,
要计算每个类别的后验概率,
选
type = "prob", 多个结果变量为.pred_{类别水平名}; - 若因变量为数值型,
要计算期望值的置信区间,
选
type = "conf_int", 结果变量为.pred_lower和.pred_upper; - 若因变量为数值型,
要计算个别值的预测区间,
选
type = "pred_int", 结果变量也是.pred_lower和.pred_upper。
比如, 计算线性回归模型在部分测试集上的预测值:
## # A tibble: 5 × 1
## .pred
## <dbl>
## 1 5.29
## 2 5.29
## 3 5.24
## 4 5.24
## 5 5.23结果是一个数据框,
其中用变量.pred保存因变量的点预测值,
各行与输入数据的各行次序一一对应。
可以用dplyr::bind_cols()将预测结果与输入的预测用数据进行横向合并,
如:
## # A tibble: 5 × 1
## .pred
## <dbl>
## 1 5.29
## 2 5.29
## 3 5.24
## 4 5.24
## 5 5.23ames_test_sm |>
select(Sale_Price) |>
bind_cols(predict(
lm_form_fit,
ames_test_sm )) |>
bind_cols(predict(
lm_form_fit,
ames_test_sm,
type = "pred_int" ))## # A tibble: 5 × 4
## Sale_Price .pred .pred_lower .pred_upper
## <dbl> <dbl> <dbl> <dbl>
## 1 5.28 5.29 4.98 5.60
## 2 5.29 5.29 4.98 5.60
## 3 5.26 5.24 4.93 5.56
## 4 4.94 5.24 4.93 5.55
## 5 5.11 5.23 4.92 5.55这样统一的预测界面的好处是, 对同一个问题, 使用不同的统计方法和计算引擎, 输出结果格式是相同的, 只要在指定模型时选择不同的统计方法和计算引擎, 后续的预报代码不变。 例如, 用树回归作预测:
tree_model <-
decision_tree(min_n = 2) |>
set_engine("rpart") |>
set_mode("regression")
tree_fit <- tree_model |>
fit(
Sale_Price ~ Longitude + Latitude,
data = ames_train )
ames_test_sm |>
select(Sale_Price) |>
bind_cols(predict(
tree_fit, ames_test_sm ))## # A tibble: 5 × 2
## Sale_Price .pred
## <dbl> <dbl>
## 1 5.28 5.31
## 2 5.29 5.31
## 3 5.26 5.15
## 4 4.94 5.15
## 5 5.11 5.1546.3 用workflow管理建模流程
一个统计建模(统计学习)过程, 不仅仅是指定模型并估计参数的问题, 还包括自变量选择、缺失值插补、变量变换、判别阈值选择等问题, 称整个过程为一个工作流程(workflow)。 其它同类软件也有类似概念, 比如Python和Spark中有流水线(pipeline)的概念。 工作流程将基于训练集产生最优模型的过程完整地封装在一起管理, 也有利于管理研究项目。
46.3.1 工作流程基础
工作流程用来管理建模估计、建模前预处理、建模后的调整。
使用线性回归建模的简单例子如下。 工作流程至少需要指定一个模型。
然后可以产生初始的工作流程:
## ══ Workflow ════════════════════════════════════════════════════════════════════
## Preprocessor: None
## Model: linear_reg()
##
## ── Model ───────────────────────────────────────────────────────────────────────
## Linear Regression Model Specification (regression)
##
## Computational engine: lm这里显示没有预处理步骤。 最简单的一种预处理步骤, 是指定一个回归公式:
## ══ Workflow ════════════════════════════════════════════════════════════════════
## Preprocessor: Formula
## Model: linear_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## Sale_Price ~ Longitude + Latitude
##
## ── Model ───────────────────────────────────────────────────────────────────────
## Linear Regression Model Specification (regression)
##
## Computational engine: lm上面的lm_wflow是一个workflow对象,
这种对象支持用fit()函数拟合模型:
## ══ Workflow [trained] ══════════════════════════════════════════════════════════
## Preprocessor: Formula
## Model: linear_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## Sale_Price ~ Longitude + Latitude
##
## ── Model ───────────────────────────────────────────────────────────────────────
##
## Call:
## stats::lm(formula = ..y ~ ., data = data)
##
## Coefficients:
## (Intercept) Longitude Latitude
## -301.337 -1.986 2.869注意lm_fit仍是一个workflow对象,
但包含了拟合结果,
也可以当做拟合结果使用,
比如用predict()产生预测,
用broom::tidy()产生参数估计数据框:
## # A tibble: 3 × 5
## term estimate std.error statistic p.value
## <chr> <dbl> <dbl> <dbl> <dbl>
## 1 (Intercept) -301. 14.3 -21.1 1.05e-90
## 2 Longitude -1.99 0.130 -15.3 1.51e-50
## 3 Latitude 2.87 0.177 16.2 2.57e-56## # A tibble: 5 × 1
## .pred
## <dbl>
## 1 5.29
## 2 5.29
## 3 5.24
## 4 5.24
## 5 5.23工作流程对象, 包括已经拟合的工作流程对象, 其中的模型和预处理内容都可以删除或者更改,例如:
## ══ Workflow ════════════════════════════════════════════════════════════════════
## Preprocessor: Formula
## Model: linear_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## Sale_Price ~ Longitude
##
## ── Model ───────────────────────────────────────────────────────────────────────
## Linear Regression Model Specification (regression)
##
## Computational engine: lm注意修改后的对象不包含拟合结果, 需要重新拟合。
46.3.2 指定变量
除了可以用add_formula()指定因变量和自变量,
还可以用add_variables()指定因变量和自变量,
指定变量集合的格式可以用tidyverse系统的tidyselect选择变量方式(参见23.8)。
如:
lm_wflow <- workflow() |>
add_model(lm_model) |>
add_variables(
outcome = Sale_Price,
predictors = c(Longtitude, Latitude) )选择自变量的例子还有:
如果指定的自变量中还包含了因变量, 因变量会自动忽略, 这样, 可以用如下方式指定所有其它变量作为自变量:
在自变量个数很多时,
tidyselect的指定变量方法可以更灵活地指定自变量集合,
而公式界面只能一一列举自变量,
或者选择使用所有除因变量以外的自变量。
但是,
add_variables()仅将选中的变量保存在数据框中传送给计算引擎,
不进行变换。
如果某些计算引擎需要用户自己将取属性值的自变量转换为哑变量,
则add_variables()不适用,
需要使用食谱(recipe)或者公式来指定因变量和自变量。
食谱是tidymodels提供的更强大的预处理功能。
46.3.3 工作流程中公式的作用
工作流程中用add_formula()添加公式,
可以用来指定因变量、自变量集合,
并在计算引擎需要时进行适当的变换,
比如将取属性值的自变量转换为哑变量,
将原始变量进行某些函数变换(如log(x))或计算(如I((x-100)^2)),
或产生交叉项,等等。
但是,
不同的扩展包和统计学习方法对变换的要求不同,如:
- 基于树的方法对取属性值的自变量并不转换为哑变量;
- 某些特殊统计模型在公式中可能使用特殊的函数名说明变量用途,
比如生存分析模型可以用
strata(site)说明site变量用于分层, 而不是当做一个普通的自变量, 所以不会估计它的分类别的截距项; - 某些特殊统计模型推广了公式,
比如在线性混合效应模型中,
用
y ~ x | id指定id作为独立观测单位的标识, 同一id值的多个观测之间是相关的。
工作流程如何利用添加的公式呢? 工作流程对公式的解释依赖于具体的模型也依赖于计算引擎(所利用的扩展包), 所以工作流程力图模拟模型需要的数据变换, 如果无法实现, 就不对公式作任何改变。 下面举例说明。
46.3.3.1 树模型的公式转换
parsnip包对于树模型能够理解计算引擎对公式的不同理解, 比如, ranger和randomForest包要求不要将取属性值的变量转换为哑变量, 而xgboost包要求进行转换, 另一个C5.0引擎则不需要转换。 parsnip对于能支持的这些计算引擎(调用的扩展包)都可以将公式按照计算引擎的特殊要求进行适当处理。
46.3.3.2 公式内的特殊语法
有些特殊模型使用特殊的公式语法, 例如lme4包建立线性混合模型, 程序例如:
这时,
对每个受试者会拟合年龄项的不同截距项和不同斜率项。
基本R不支持这样的公式,比如model.matrix()函数不能基于这样的公式产生线性模型设计阵。
在工作流程中解决这个问题的办法在add_model()中同时指定对应的公式,
而用到的变量则使用add_variables()原样传递,
如:
## Warning: package 'multilevelmod' was built under R version 4.2.3data(Orthodont, package = "nlme")
multilevel_spec <- linear_reg() |>
set_engine("lmer")
multilevel_workflow <- workflow() |>
add_variables(
outcome = distance,
predictors = c(Sex, age, Subject) ) |>
add_model(
multilevel_spec,
formula = distance ~ Sex + (age | Subject) )
multilevel_fit <- fit(
multilevel_workflow,
data = Orthodont )
multilevel_fit## ══ Workflow [trained] ══════════════════════════════════════════════════════════
## Preprocessor: Variables
## Model: linear_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## Outcomes: distance
## Predictors: c(Sex, age, Subject)
##
## ── Model ───────────────────────────────────────────────────────────────────────
## Linear mixed model fit by REML ['lmerMod']
## Formula: distance ~ Sex + (age | Subject)
## Data: data
## REML criterion at convergence: 471.1635
## Random effects:
## Groups Name Std.Dev. Corr
## Subject (Intercept) 7.3912
## age 0.6943 -0.97
## Residual 1.3100
## Number of obs: 108, groups: Subject, 27
## Fixed Effects:
## (Intercept) SexFemale
## 24.517 -2.14546.3.3.3 公式内的特殊函数
上述办法也能解决公式中有特定扩展包支持的特殊公式的问题。
比如,
在生存分析中,
可以用strata(site)表示site用来分层,
工作流程程序例如:
## Warning: package 'censored' was built under R version 4.2.3## Loading required package: survivalparametric_spec <- survival_reg()
parametric_workflow <- workflow() |>
add_variables(
outcome = c(fustat, futime),
predictors = c(age, rx) ) |>
add_model(
parametric_spec,
formula = Surv(futime, fustat) ~ age + strata(rx) )
parametric_fit <- fit(
parametric_workflow,
data = ovarian )
parametric_fit## ══ Workflow [trained] ══════════════════════════════════════════════════════════
## Preprocessor: Variables
## Model: survival_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## Outcomes: c(fustat, futime)
## Predictors: c(age, rx)
##
## ── Model ───────────────────────────────────────────────────────────────────────
## Call:
## survival::survreg(formula = Surv(futime, fustat) ~ age + strata(rx),
## data = data, model = TRUE)
##
## Coefficients:
## (Intercept) age
## 12.8734120 -0.1033569
##
## Scale:
## rx=1 rx=2
## 0.7695509 0.4703602
##
## Loglik(model)= -89.4 Loglik(intercept only)= -97.1
## Chisq= 15.36 on 1 degrees of freedom, p= 8.88e-05
## n= 2646.3.4 批量建立工作流程
统计建模的过程, 一般都需要比较多个模型, 有时需要比较不同的变量子集。 workflowsets包可以指定预处理器列表和模型列表, 然后生成一批工作流程, 最后设法比较拟合结果。
以Ames数据为例, 考虑使用待售房屋方位的不同方式, 产生多个工作流程比较效果。 首先产生不同的使用房屋方位的列表:
location <- list(
longitude = Sale_Price ~ Longitude,
latitude = Sale_Price ~ Latitude,
coords = Sale_Price ~ Longitude + Latitude,
neighborhood = Sale_Price ~ Neighborhood )这些方式, 可以与不同模型交叉产生多个工作流程。 下面的例子仅使用了线性模型:
library(workflowsets)
location_models <- workflow_set(
preproc = location,
models = list(lm = lm_model) )
location_models## # A workflow set/tibble: 4 × 4
## wflow_id info option result
## <chr> <list> <list> <list>
## 1 longitude_lm <tibble [1 × 4]> <opts[0]> <list [0]>
## 2 latitude_lm <tibble [1 × 4]> <opts[0]> <list [0]>
## 3 coords_lm <tibble [1 × 4]> <opts[0]> <list [0]>
## 4 neighborhood_lm <tibble [1 × 4]> <opts[0]> <list [0]>可见,
workflow_sets()的结果是一个tibble数据框,
每一行是一个工作流程。
info列示例:
## # A tibble: 1 × 4
## workflow preproc model comment
## <list> <chr> <chr> <chr>
## 1 <workflow> formula linear_reg ""option列示例:
## an empty container for options提取某一行的工作流程示例:
## ══ Workflow ════════════════════════════════════════════════════════════════════
## Preprocessor: Formula
## Model: linear_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## Sale_Price ~ Longitude + Latitude
##
## ── Model ───────────────────────────────────────────────────────────────────────
## Linear Regression Model Specification (regression)
##
## Computational engine: lm为了比较工作流程集合中的不同模型,
一般会使用交叉验证等重抽样方法,
重抽样设置与比较结果会保存在工作流程集合数据框的result列中。
这里先不进行重抽样比较,
先在训练数据上对每个模型进行拟合。
这可以用purrr包的map()函数,
以及dplyr包的mutate在工作流程集合数据框添加拟合结果列fit:
location_models <- location_models |>
mutate(
fit = map(
info,
~ fit(
.x$workflow[[1]],
ames_train)))
location_models## # A workflow set/tibble: 4 × 5
## wflow_id info option result fit
## <chr> <list> <list> <list> <list>
## 1 longitude_lm <tibble [1 × 4]> <opts[0]> <list [0]> <workflow>
## 2 latitude_lm <tibble [1 × 4]> <opts[0]> <list [0]> <workflow>
## 3 coords_lm <tibble [1 × 4]> <opts[0]> <list [0]> <workflow>
## 4 neighborhood_lm <tibble [1 × 4]> <opts[0]> <list [0]> <workflow>在上面的程序中,
mutate()输入location_models中的info列,
这是一个列表,
列表的每个元素是一个工作流程的信息,
每个工作流程信息是一个仅有一行的tibble数据框,
其中的workflow列是一个仅有一行的列表,
元素为工作流程,
但必须用.x$workflow[[1]]这样的格式取出,
而不是用.x$workflow取出。
.x是map()函数中用公式语法定义处理函数时的自变量名。
第一个工作流程的拟合结果:
## ══ Workflow [trained] ══════════════════════════════════════════════════════════
## Preprocessor: Formula
## Model: linear_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## Sale_Price ~ Longitude
##
## ── Model ───────────────────────────────────────────────────────────────────────
##
## Call:
## stats::lm(formula = ..y ~ ., data = data)
##
## Coefficients:
## (Intercept) Longitude
## -179.412 -1.972虽然使用mutate()和map()批量拟合模型很方便,
但是后面会给出更有用的批量拟合、比较模型的办法。
46.3.5 使用测试集
在一般的统计学习任务中, 测试集不应该用来比较不同模型, 而是在选择了最优模型后, 用来给出其预测性能的评判指标。 比较不同模型应该仅使用训练集。
现在假设已经有了最优模型,
比如是工作流程lm_wflow的结果,
则可以使用last_fit()在测试集上进行性能测试,
这时,
模型估计利用了整个训练集,
在测试集上测试其性能:
lm_model <- linear_reg() |>
set_engine("lm")
lm_wflow <- workflow() |>
add_model(lm_model) |>
add_formula(
Sale_Price ~ Longitude + Latitude )
final_lm_res <- last_fit(lm_wflow, ames_split)
final_lm_res## # Resampling results
## # Manual resampling
## # A tibble: 1 × 6
## splits id .metrics .notes .predictions .workflow
## <list> <chr> <list> <list> <list> <list>
## 1 <split [2344/586]> train/test split <tibble> <tibble> <tibble> <workflow>提取所使用的工作流程:
## ══ Workflow [trained] ══════════════════════════════════════════════════════════
## Preprocessor: Formula
## Model: linear_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## Sale_Price ~ Longitude + Latitude
##
## ── Model ───────────────────────────────────────────────────────────────────────
##
## Call:
## stats::lm(formula = ..y ~ ., data = data)
##
## Coefficients:
## (Intercept) Longitude Latitude
## -301.337 -1.986 2.869提取测试性能指标:
## # A tibble: 2 × 4
## .metric .estimator .estimate .config
## <chr> <chr> <dbl> <chr>
## 1 rmse standard 0.167 Preprocessor1_Model1
## 2 rsq standard 0.165 Preprocessor1_Model1提取在测试集上的预测结果, 仅显示前几项:
## # A tibble: 5 × 5
## id .pred .row Sale_Price .config
## <chr> <dbl> <int> <dbl> <chr>
## 1 train/test split 5.29 5 5.28 Preprocessor1_Model1
## 2 train/test split 5.29 6 5.29 Preprocessor1_Model1
## 3 train/test split 5.24 29 5.26 Preprocessor1_Model1
## 4 train/test split 5.24 32 4.94 Preprocessor1_Model1
## 5 train/test split 5.23 33 5.11 Preprocessor1_Model146.4 用recipe进行特征工程
训练数据集中的原始变量并不一定是最适合用来预报因变量的, 对原始变量进行一些变换、重新编码, 或者利用多个变量计算有意义的新变量, 都可能会改进模型的效果。 这样的做法称为“特征工程”(feature engineering)。 特征工程还可以筛选有作用的自变量, 进行降维, 插补缺失值, 将有偏分布转换成对称分布, 等等。 有些模型依赖于简单的欧式距离, 这时各个自变量应该进行标准化, 否则在计算距离时各个变量的影响会有很大的差异。 不同的模型对自变量形式要求也不同, 比如, 基于树的方法不要求分类变量转换为哑变量, 而回归方法则一般要把分类变量转换为哑变量表示(机器学习中称为独热(one-hot)编码)。
recipe包可以把多步特征工程和预处理操作整合为一个对象, 可以将其应用于多个数据集。 这是tidymodels扩展包组合的核心成员之一。
46.4.1 Ames数据集示例
已ames数据集为例, 进行简单的特征工程示例。 仅考虑如下几个自变量:
neighborhood, 分类变量,训练集中共有29个不同取值;Gr_Liv_Area,数值型变量,总的达标居住面积;Year_Built,建造年份;Bldg_Type,房屋类型,分类变量,可取:OneFam;TwoFmCon;Duplex;Twnhs;TwnhsE。
因变量Sale_Price已经变换成了常用对数值。
假设使用lm()函数计算线性回归:
该函数会自动将自变量提取出来组成为设计阵,
这会把分类变量(neighborhood和Bldg_Type)都变成哑变量形式,
变量Gr_Liv_Area会按公式要求用常用对数转换。
如果使用回归结果进行预测,
预测所用的新数据也会进行相同的转换。
一个食谱(recipe)用step_*函数指定类似的预处理步骤,
但暂不进行实际的计算。
例如,
下面的程序可以产生类似于上述lm()对数据进行的变量,
但仅规定变换操作,
暂不进行实际变换:
library(tidymodels)
tidymodels_prefer()
simple_ames <-
recipe(
Sale_Price ~ Neighborhood + Gr_Liv_Area
+ Year_Built + Bldg_Type,
data = ames_train) |>
step_log(Gr_Liv_Area, base = 10) |>
step_dummy(all_nominal_predictors())
simple_ames## ## ── Recipe ──────────────────────────────────────────────────────────────────────## ## ── Inputs## Number of variables by role## outcome: 1
## predictor: 4## ## ── Operations## • Log transformation on: Gr_Liv_Area## • Dummy variables from: all_nominal_predictors()上面的程序中,
用recipe添加了因变量和自变量集合(没有指定对自变量的变换),
以及训练数据集。
然后,用管道操作,
给recipe的结果用step_log对变量Gr_Liv_Area指定了作常用对数变换,
用step_dummy指定了将已选定的自变量集合中所有分类变量转换为哑变量形式。
在recipe环境下,
用all_nominal_predictors()表示所有分类自变量变量,
用all_numeric_predictors()表示所有数值型自变量,
用all_predictors()表示所有自变量,
用all_outcomes()表示所有因变量。
step_*函数中可以指定多个变量,
只要原样写变量名(不用撇号保护)并用逗号分隔。
在recipe()中用data =指定训练数据集,
这时仅从数据确定变量集合与变量类型,
但不进行实际的变量转换。
使用食谱处理变量变换, 而不是直接使用R的公式界面, 或者直接指定变量, 有如下优势:
- 如果使用了不同的模型,这些变换可以在不同模型之间共用;
- 食谱能做的变换比R的公式界面功能更强;
- 使用更方便,比如可以使用
all_nominal_predictors()表示所有分类自变量变量, 而公式中则没有这样的方法; - 对数据要进行的相同的预处理可以存放在一个单一的R对象中, 而不是在源程序中多次重复,甚至于在多个源文件中重复。
46.4.2 使用食谱作为预处理
数据预处理是工作流程的一部分, 在工作流程中加入食谱, 是更灵活的数据预处理指定方式。
工作流程只能有一种预处理方式,
不能在使用add_formula()或add_variables()后再使用食谱。
lm_model <- linear_reg() |>
set_engine("lm")
lm_wflow <- workflow() |>
add_model(lm_model) |>
add_recipe(simple_ames)
lm_wflow## ══ Workflow ════════════════════════════════════════════════════════════════════
## Preprocessor: Recipe
## Model: linear_reg()
##
## ── Preprocessor ────────────────────────────────────────────────────────────────
## 2 Recipe Steps
##
## • step_log()
## • step_dummy()
##
## ── Model ───────────────────────────────────────────────────────────────────────
## Linear Regression Model Specification (regression)
##
## Computational engine: lm上面的食谱(预处理)仅进行了设置,
而没有进行实际的转换。
用fit()函数,
则可以将工作流程中的预处理进行实际计算,
并拟合模型:
在fit()函数中使用工作流程,
就会调用工作流程中的食谱,
进行实际的变量变换。
这些变换仅针对训练集。
用predict()对测试集的前三个观测计算预测值:
## Warning in predict.lm(object = object$fit, newdata = new_data, type =
## "response"): prediction from a rank-deficient fit may be misleading## # A tibble: 3 × 1
## .pred
## <dbl>
## 1 5.28
## 2 5.28
## 3 5.22在用predict()对测试集进行预测时,
也会在测试集上调用食谱进行预处理,
但这些预处理的参数还是使用从训练集得到的参数,
比如,
如果预处理是对变量作标准化,
即减去变量均值后除以变量标准差,
均值和标准差都是在训练集上计算,
用predict()对测试集进行预测时的变量标准化仍使用训练集上计算的变量均值和标准差。
可以用extract_recipe()加选项estimated = TRUE从拟合结果中提取已计算的预处理结果,如:
## ## ── Recipe ──────────────────────────────────────────────────────────────────────## ## ── Inputs## Number of variables by role## outcome: 1
## predictor: 4## ## ── Training information## Training data contained 2344 data points and no incomplete rows.## ## ── Operations## • Log transformation on: Gr_Liv_Area | Trained## • Dummy variables from: Neighborhood, Bldg_Type | Trainedextract_fit_parsnip()从拟合结果中提取与模型和计算引擎有关的方面,
可以获得模型估计结果,
用tidy()函数可以提取整洁形式的模型参数估计:
| term | estimate | std.error | statistic | p.value |
|---|---|---|---|---|
| (Intercept) | -0.6494257 | 0.2249441 | -2.887054 | 0.0039248 |
| Gr_Liv_Area | 0.6160497 | 0.0138745 | 44.401726 | 0.0000000 |
| Year_Built | 0.0019915 | 0.0001134 | 17.554954 | 0.0000000 |
| Neighborhood_College_Creek | 0.0159125 | 0.0081121 | 1.961565 | 0.0499331 |
| Neighborhood_Old_Town | -0.0290021 | 0.0082649 | -3.509056 | 0.0004583 |
46.4.3 典型的预处理
recipe包的step_*函数提供了各种常见的预处理能力,
这里举例说明。
46.4.3.1 将类别变量转码为数值型
step_dummy()可以将指定的类别变量(名义型、属性值)转换为哑变量编码。
对保存成R因子类型的类别变量,
也可以作一些水平的变化,
比如,
用step_unknown()将指定的类别变量的缺失值转换成一个因子水平值,
用step_novel()为指定的类别变量预留一个原来没有但将来可能出现的因子水平值,
step_other()可以统计各水平的频数,
将低频的水平值合并到一个other类别,
等等。
如:
simple_ames <-
recipe(Sale_Price ~ Neighborhood
+ Gr_Liv_Area + Year_Built + Bldg_Type,
data = ames_train) |>
step_log(Gr_Liv_Area, base = 10) |>
step_other(Neighborhood, threshold = 0.01) |>
step_dummy(all_nominal_predictors())其中的step_other()调用就将Neighborhood中总百分比小于\(1\%\)的低频取值合并成一个other类别。
注意:多数的模型需要将类别变量转换为数值型变量, 一般是转换为哑变量(参见33.10), 但基于树的模型、基于规则的模型、朴素贝叶斯模型不需要转换。
step_dummy()进行标准的哑变量变换,
比如,
Neighborhood有5个类别,
就会转换成4个哑变量,
对应于5个类别中的后面4个类别,
命名格式是“变量名_类别值”。
每个哑变量都取值0或者1,
但每个观测仅有一个哑变量取1,
取值1的哑变量对应于相应的类别,
当4个哑变量全取0时对应于基线类别,
默认是因子的第一个水平。
可以用step_dummy()中的onehot = TRUE选项要求生成与类别个数相同的哑变量。
除了最常见的哑变量编码, recipes也支持其它的类别变量编码方法。
46.4.3.2 交互作用项
在R的公式中用a:b表示a和b的交互作用效应。
如果a和b都是数值型的,
则交互作用效应等于两者的乘积。
如果a是分类的,
b是数值型的,
则交互作用效应是a的每个类别分别计算b的系数(斜率)。
如果a和b都是分类的,
则对a, b的所有组合建立哑变量编码。
recipes中用step_interact()指定交互作用项,如:
simple_ames <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area
+ Year_Built + Bldg_Type,
data = ames_train) |>
step_log(Gr_Liv_Area, base = 10) |>
step_other(Neighborhood, threshold = 0.01) |>
step_dummy(all_nominal_predictors()) |>
# 注意下面的Gr_Liv_Area是已经做完对数变换的结果
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") )上面的程序中step_interact()建立了数值型的Gr_Liv_Area的对数变换,
与已经做过哑变量编码的Bldg_Type之间的交互作用效应。
由变量a, b产生的交互作用效应变量命名为a_x_b的格式。
如果需要添加多个交互作用效应,
只要在公式的右侧用+号连接。
注意建立交互作用效应时, 如果有类别变量参与, 应该预先进行哑变量编码。 基本R的公式界面没有这样的要求。
46.4.3.3 样条函数项
在基本R的公式中可以用ns(x, df=3)这样的方式增加变量x的样条函数变换作为自变量。
这种变换将一列数值型变量x的值替换成若干个样条函数值的列,
提供灵活的非线性拟合能力。
自由度越大, 越容易产生过度拟合。 不同的自由度的拟合样例:
library(patchwork)
library(splines)
plot_smoother <- function(deg_free) {
ggplot(ames_train, aes(
x = Latitude, y = 10^Sale_Price)) +
geom_point(alpha = .2) +
scale_y_log10() +
geom_smooth(
method = lm,
formula = y ~ ns(x, df = deg_free),
color = "lightblue",
se = FALSE
) +
labs(
title = paste(deg_free, "Spline Terms"),
y = "Sale Price (USD)")
}
( plot_smoother(2) + plot_smoother(5) ) /
( plot_smoother(20) + plot_smoother(100) )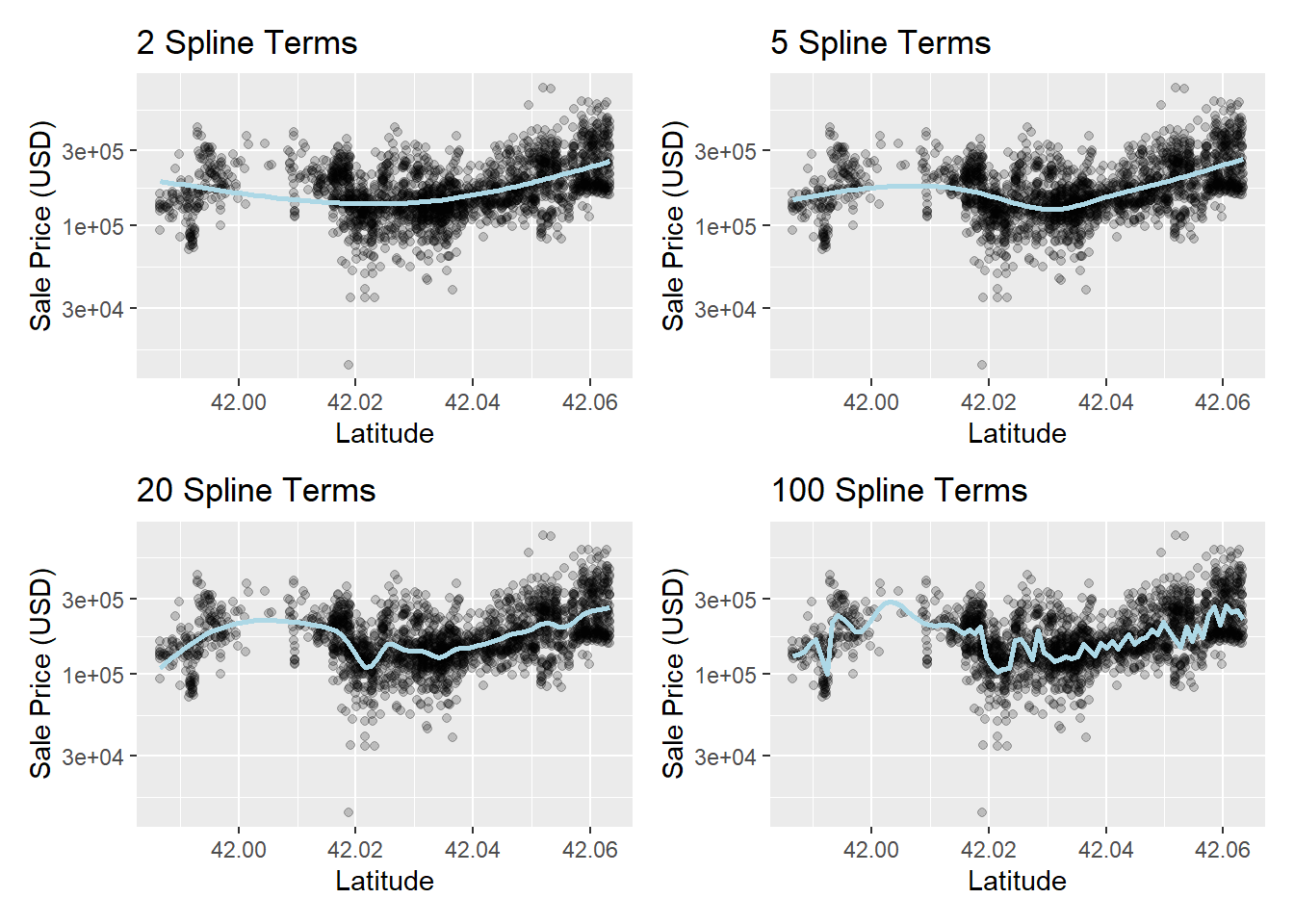
用recipes添加样条变换项的例子:
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area
+ Year_Built + Bldg_Type + Latitude,
data = ames_train) |>
step_log(Gr_Liv_Area, base = 10) |>
step_other(Neighborhood, threshold = 0.01) |>
step_dummy(all_nominal_predictors()) |>
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") ) |>
step_ns(Latitude, deg_free = 20)46.4.3.4 特征提取
预处理经常需要根据旧有变量计算产生对预测因变量更有用的新变量。 比如, 主成分分析(PCA)就是一种消除旧有变量中的相关性并获得压缩结果的方法。 例如, ames数据中与面积大小有关的变量相关性强, 可以进行压缩, 程序如:
其中用了matches()指定变量名符合特定模式的变量子集。
如果各个变量量纲不同,
应该先用step_normalize()将变量转换到相近的取值范围。
除了主成分分析以外, recipes还支持其它一些变换方法。
46.4.3.5 对观测行进行抽样
在分类问题中, 如果两个类别的比例相差很大, 很容易忽略频率较少的类。 为此可以对训练样本的观测进行抽样, 产生新的训练样本:
- 可以减数抽样, 保持少数派观测不变, 对多数派样本仅抽取其中一部分;
- 可以增殖抽样, 保持多数派观测不变, 对少数派进行抽样增加, 增加方式可能是简单地重复, 也可能是产生与少数派表现相同但不同的样本。
- 可以对多数派抽取一部分, 对少数派增加样品。
themis包能够进行这些操作, 比如, 减数抽样可用:
这些对样本行的增删应该仅对训练样本进行,
测试样本和其它保留的样本不应该进行这些操作。
这些函数设置了一个skip参数,
默认为TRUE,
就是规定这样的原则。
还有一些操作样本行的函数,
如step_filter(),
step_sample(),
step_slice(),
step_arrange()。
46.4.4 对新数据应避免的预处理
如果需要对因变量进行变换, 不应该作为预处理步骤规定在食谱中, 这是因为在使用测试集时, 测试集中可以没有因变量存在。
如前所述,
如果对数据行进行了一些操作,
比如减数抽样,
应该仅对训练集进行,
但是测试集上不应该进行这些操作。
这样的行操作函数提供了skip = TRUE默认选项,
就是要求在predict()时忽略行操作预处理步骤。
当然,
fit()函数不会忽略这样的步骤。
如下的行操作函数仅对训练集有效:
step_adasyn(),
step_bsmote(),
step_downsample(),
step_filter(),
step_naomit(),
step_nearmiss(),
step_rose(),
step_sample(),
step_slice(),
step_smote(),
step_smotenc(),
step_tomek(),
step_upsample()。
46.4.5 食谱的整洁表示
可以用tidy()函数将食谱转换为数据框,
如:
ames_rec <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area
+ Year_Built + Bldg_Type
+ Latitude + Longitude,
data = ames_train) |>
step_log(Gr_Liv_Area, base = 10) |>
step_other(Neighborhood, threshold = 0.01,
id = "nei_other") |>
step_dummy(all_nominal_predictors()) |>
step_interact(
~ Gr_Liv_Area:starts_with("Bldg_Type_") ) |>
step_ns(Latitude, Longitude, deg_free = 20)
tidy(ames_rec) |> knitr::kable()| number | operation | type | trained | skip | id |
|---|---|---|---|---|---|
| 1 | step | log | FALSE | FALSE | log_WPwWU |
| 2 | step | other | FALSE | FALSE | nei_other |
| 3 | step | dummy | FALSE | FALSE | dummy_Z4Mwx |
| 4 | step | interact | FALSE | FALSE | interact_pPAiA |
| 5 | step | ns | FALSE | FALSE | ns_LGEm4 |
数据集中的id是自动生成的,
可以在step_*函数中用id =认为指定,
如上面的step_other()调用。
下面利用上述预处理重新拟合:
lm_wflow <-
workflow() |>
add_model(lm_model) |>
add_recipe(ames_rec)
lm_fit <- fit(lm_wflow, ames_train)下面提取拟合过的step_other()预处理步的信息,
这时,
上面指定的id就可以用来从所有预处理步中定位:
estimated_recipe <-
lm_fit %>%
extract_recipe(estimated = TRUE)
tidy(estimated_recipe, id = "nei_other")## # A tibble: 20 × 3
## terms retained id
## <chr> <chr> <chr>
## 1 Neighborhood North_Ames nei_other
## 2 Neighborhood College_Creek nei_other
## 3 Neighborhood Old_Town nei_other
## 4 Neighborhood Edwards nei_other
## 5 Neighborhood Somerset nei_other
## 6 Neighborhood Northridge_Heights nei_other
## 7 Neighborhood Gilbert nei_other
## 8 Neighborhood Sawyer nei_other
## 9 Neighborhood Northwest_Ames nei_other
## 10 Neighborhood Sawyer_West nei_other
## 11 Neighborhood Mitchell nei_other
## 12 Neighborhood Brookside nei_other
## 13 Neighborhood Crawford nei_other
## 14 Neighborhood Iowa_DOT_and_Rail_Road nei_other
## 15 Neighborhood Timberland nei_other
## 16 Neighborhood Northridge nei_other
## 17 Neighborhood Stone_Brook nei_other
## 18 Neighborhood South_and_West_of_Iowa_State_University nei_other
## 19 Neighborhood Clear_Creek nei_other
## 20 Neighborhood Meadow_Village nei_other这个拟合过的预处理步显示结果包含了没有被合并的类别的列表。
tidy()函数除了用id定位某一个预处理步,
也可以用number =指定预处理步的序号。
对食谱调用tidy()函数,
可以选择某个预处理步,
显示与该预处理步有关的信息。
46.4.6 变量的角色
在公式中出现的变量, 基本的角色是自变量(predictor)和因变量(outcome)。 还可以有其它角色。
比如,
ames数据集的每一行可以有一个地址,
这虽然不是自变量也不是因变量,
但可以用来定位感兴趣的观测。
可以用add_role()、remove_role()、update_role()改变变量的角色。
如:
角色名称可以是任何字符串。
一个数据集变量也可以有多重角色,
可以用add_role()在已有角色时额外添加角色。
在数据集中保存非自变量、因变量角色的变量列, 好处是在进行行操作、随机抽样操作时, 可以同步地对这些列进行相同的操作, 如果使用数据集外部的一个变量保存这些信息(如地址), 就不能自动跟随这些行操作。
46.4.7 更多预处理步骤
recipes和相关的包提供了上百个预处理方法, 参见https://www.tidymodels.org/find/。 还可以自己定义预处理步, 参见https://www.tidymodels.org/learn/develop/recipes/。
46.5 模型效果的评价指标计算
建模需要能够定量地评估模型效果, 用来比较不同的模型、对模型进行参数调优、更好地理解模型。 这里主要使用验证数据集来评估模型效果。 作为示例, 这一节将测试数据集当作验证数据集来使用, 但实际建模时应该使用单独的验证数据集, 或者使用随机抽样方法进行交叉验证。
选择什么样的定量度量来评估模型, 对最后选择的模型有关键的影响。 比如, 在回归分析中, 一般使用最小二乘估计, 这最小化了均方误差, 使得预测误差最小; 如果参数估计时最大化复相关系数平方\(R^2\), 这实际上是最大化了因变量的真实值和拟合值的相关系数, 一般是不合理的做法。
yardstick是tidymodels扩展包集合中的核心组成包, 提供了各种模型评估度量。
46.5.1 模型评估与统计推断
本章对建模更强调模型的预测能力, 即使建模主要是为了进行统计推断, 其预测能力的评估也可以从另一侧面验证模型的可用性。
常见的推断性统计模型应用往往是不断在不同模型设定之间进行比较检验, 直到不能继续增加或减少解释项。 但是, 要注意这样还主要是一个拟合问题, 不管模型拟合的有多么好, 也不能说明这个模型就是客观规律的良好近似, 这是拟合问题的固有缺陷。 这时, 借助于交叉验证等方法进行预测能力的评估, 可以帮助我们认识模型的可信性。
46.5.2 回归问题的评估
在完成模型拟合后,
可以产生验证集或测试集上的预测结果,
将预测结果保存入数据集,
然后,
yardstick包的评估指标计算函数有统一的界面:
如果函数名为f,
保存预测结果的数据集为data,
保存因变量真实值的列为truth,
则计算评估指标的调用格式为
其中的...参数用来定位数据中的预测值。
下面用ames数据集举例说明,
先拟合模型:
ames_rec <-
recipe(Sale_Price ~ Neighborhood + Gr_Liv_Area
+ Year_Built + Bldg_Type
+ Latitude + Longitude,
data = ames_train) |>
step_log(Gr_Liv_Area, base = 10) |>
step_other(Neighborhood, threshold = 0.01) |>
step_dummy(all_nominal_predictors()) |>
step_interact( ~ Gr_Liv_Area:starts_with("Bldg_Type_") ) |>
step_ns(Latitude, Longitude, deg_free = 20)
lm_model <- linear_reg() |> set_engine("lm")
lm_wflow <-
workflow() |>
add_model(lm_model) |>
add_recipe(ames_rec)
lm_fit <- fit(lm_wflow, ames_train)为了演示,
这里用ames_test当作验证数据集,
正常情况下应该单独生成验证数据集或者使用训练集的随机抽样交叉验证。
在验证数据集上进行预测,
产生因变量的预测值:
ames_test_res <- predict(
lm_fit,
new_data = ames_test |> select(-Sale_Price))
ames_test_res |> slice(1:5)## # A tibble: 5 × 1
## .pred
## <dbl>
## 1 5.28
## 2 5.27
## 3 5.22
## 4 5.08
## 5 5.14回归结果的点预测值,
在保存预测结果的数据框中命名为.pred。
可以将预测结果与验证集的原始数据中的因变量真实值一一对应起来保存:
ames_test_res <- ames_test |>
select(Sale_Price) |>
bind_cols(ames_test_res)
ames_test_res |> slice(1:5)## # A tibble: 5 × 2
## Sale_Price .pred
## <dbl> <dbl>
## 1 5.28 5.28
## 2 5.29 5.27
## 3 5.26 5.22
## 4 4.94 5.08
## 5 5.11 5.14可以浏览这个两列的数据集, 大致地感觉预测精度。 注意因变量真实值和预测值都是对数变换结果, 在评估模型效果时, 一般使用建模时的因变量形式, 而不是转换成原始形式再评估。
可以作散点图显示两者的关系:
ggplot(ames_test_res, aes(
x = Sale_Price, y = .pred)) +
geom_abline(lty = 2) + # 没有指定a, b则默认a=0, b=1
geom_point(alpha = 0.5) +
labs(y = "Predicted Sale Price (log10)",
x = "Sale Price (log10)") +
coord_obs_pred() # 令x、y轴使用相同长度单位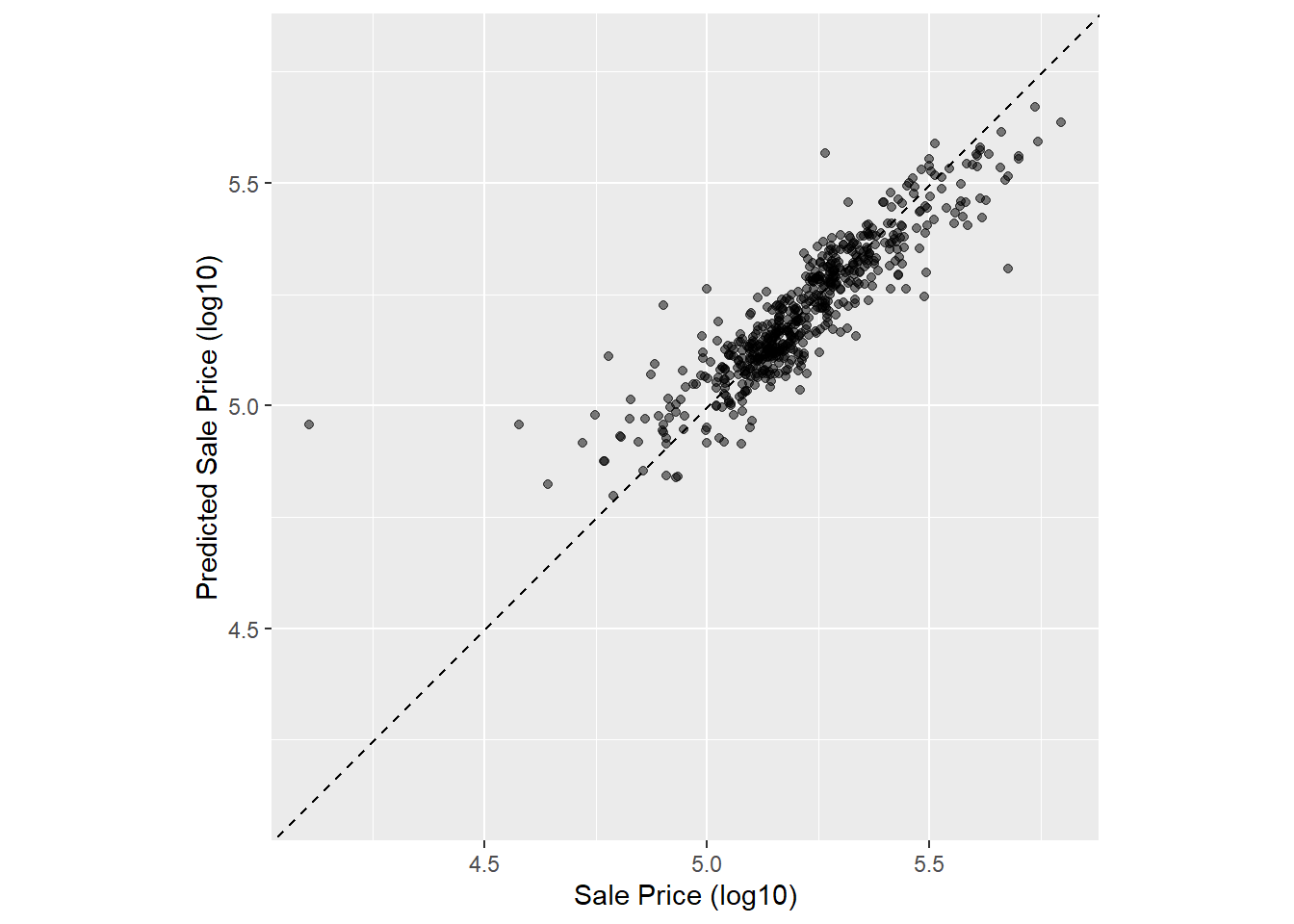
在有了保存因变量预测值和真值的数据集后,
可以用rmse()函数计算根均方误差:
\[
\text{RMSE} = \sqrt{\frac{1}{N} \sum_{i=1}^N
(y_i - \hat y_i)^2} .
\]
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 rmse standard 0.0828注意.pred不需要用撇号保护。
rmse()的如上调用方法是yardstick包中计算模型评估指标的函数的普遍坐法。
如果要同时计算多个模型评估指标,
因为每个评估指标对应于一个函数,
需要用metric_set()函数将若干个评估指标函数组合成一个评估指标集合函数,
调用这个函数就可以计算多个评估指标。
如:
ames_metrics <- metric_set(rmse, rsq, mae)
ames_metrics(
ames_test_res,
truth = Sale_Price,
estimate = .pred)## # A tibble: 3 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 rmse standard 0.0828
## 2 rsq standard 0.797
## 3 mae standard 0.0561这计算了根均方误差,\(R^2\),平均绝对误差: \[ \text{MAE} = \frac{1}{N} \sum_{i=1}^N |y_i - \hat y_i| . \] RMSE和MAE与因变量量纲相同, 度量因变量和自变量的取值接近程度; 而\(R^2\)是一个取值在\([0, 1]\)区间的数值, \(R^2\)越大, 表示拟合越好。 \(R^2\)实际是因变量真实值与拟合值的相关系数的平方。
yardstick有意地取消了修正\(R^2\)的计算功能, 因为修正\(R^2\)主要是在同一个训练集上比较模型, 而更好的做法是在不同数据上评估模型预测效果。
下面用随机森林方法对ames数据集进行预测, 并与线性模型结果进行比较:
rf_model <-
rand_forest(trees = 1000) |>
set_engine("ranger") |>
set_mode("regression")
rf_wflow <-
workflow() |>
add_model(rf_model) |>
add_formula(
Sale_Price ~ Neighborhood + Gr_Liv_Area
+ Year_Built + Bldg_Type
+ Latitude + Longitude)
# 注意上面没有将因子转换成哑变量,随机森林不需要
rf_fit <- fit(rf_wflow, ames_train)
ames_test_res_rf <- predict(
rf_fit,
new_data = ames_test |> select(-Sale_Price))
ames_test_res_rf <- ames_test |>
select(Sale_Price) |>
bind_cols(ames_test_res_rf)
ames_metrics <- metric_set(rmse, rsq, mae)
ames_metrics(
ames_test_res_rf,
truth = Sale_Price,
estimate = .pred)## # A tibble: 3 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 rmse standard 0.0749
## 2 rsq standard 0.840
## 3 mae standard 0.0480线性回归在测试集上的MAE为\(0.055\), 随机森林的MAE为\(0.051\), 随机森林略优。 但是这样的评估结果有随机性, 如果重新划分训练集、测试集, 评估结果的比较可能会改变。 更好的模型比较方法是利用再抽样技术, 见47.1。
46.5.3 二分类问题的评估
46.5.3.1 二分类问题的评估指标
二分类问题预测的评估有许多种指标, 各有侧重。
在医学检验中, 将有某种病的人正确检出的概率叫做敏感度(sensitivity), 将无病的人判为无病的概率叫做特异度(specificity)。 将这样的术语对应到统计假设检验问题: \[ H_0: \text{无病} \longleftrightarrow H_a: \text{有病} \] 第一类错误是\(H_0\)成立但拒绝\(H_0\)的错误, 概率为\(\alpha\); 第二类错误是\(H_a\)成立但承认\(H_0\)的错误, 概率为\(\beta\)。 则敏感度为\(1-\beta\)(检验功效), 特异度为\(1-\alpha\)。
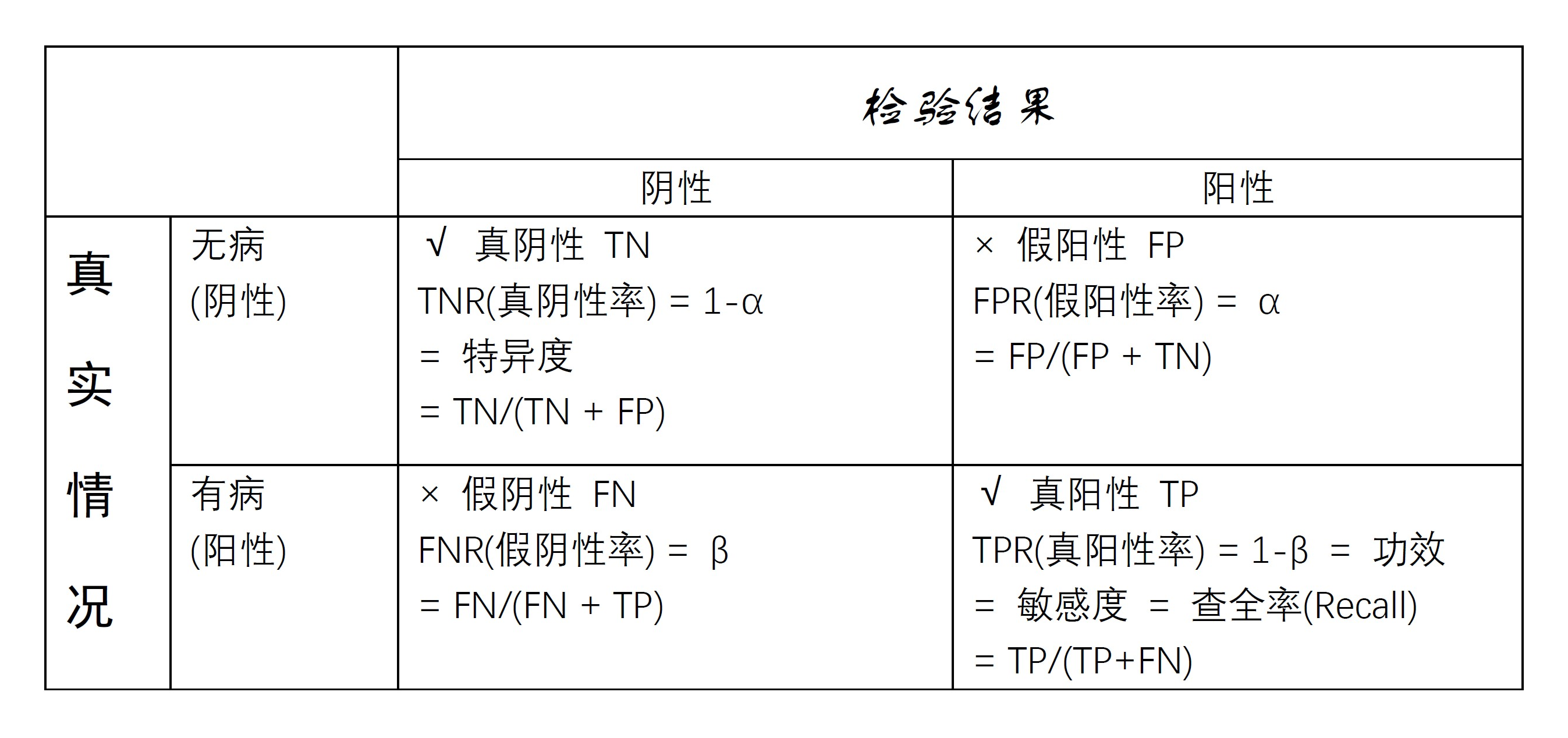
图46.1: 医学诊断四种情况表格
称\(\alpha\)为假阳性率(FPR), 等于1减去特异度; 称敏感度为真阳性率(TPR), 也称为召回率(recall)。
设图46.1中TN表示真阴性例数, 即无病判为无病个数; FN表示假阴性例数, 即有病判为无病例数; FP表示假阳性例数, 即无病判为有病例数; TP表示真阳性例数, 即有病判为有病例数。 这时,可以估计 \[\begin{aligned} \text{TPR(真阳性率)} =& 1 - \beta = \text{Sensitivity(敏感度)} = \text{Recall(查全率)} = \frac{TP}{TP + FN} ; \\ \text{FPR(假阳性率)} =& \alpha = \frac{FP}{FP + TN} ; \\ \text{TNR(真阴性率)} =& 1-\alpha = \text{Specificity(特异度)} = \frac{TN}{TN + FP}; \\ \text{FNR(假阴性率)} =& \beta = \frac{FN}{FN + TP} . \end{aligned}\]
另外, \[\begin{aligned} \text{Precision(查准率)} =& \text{Positive predictive value} = 1 - \text{FDR} = \frac{TP}{TP + FP}; \\ \text{FDR(错误发现率)} =& \frac{FP}{FP + TP} = 1 - \text{Precision}. \end{aligned}\]
F1指标是查准率与查全率的如下“调和平均”: \[ \frac{1}{\text{F1}} = \frac{1}{2} \left( \frac{1}{\text{Precision}} + \frac{1}{\text{Recall}} \right) . \] F1指标更重视将正例检出, 而忽视对反例的正确检出。
MCC(Mathew相关系数)度量\(2 \times 2\)列联表行、列变量的相关性, 与拟合优度卡方统计量等价,计算公式为: \[ \text{MCC} = \frac{\text{TN} \times \text{TP} - \text{FN} \times \text{FP} }{ \sqrt{ (\text{TP} + \text{FP}) (\text{TP} + \text{FN}) (\text{TN} + \text{FP}) (\text{TN} + \text{FN}) }} . \]
在两类的分类问题中, 将两类分别称为正例、反例, 或者阳性、阴性, 或者事件、非事件。 设自变量\(x\)得到的正例的后验概率为\(f(\boldsymbol x)\), 任意取分界值\(t\), 都可以按\(f(\boldsymbol x) > t\)和\(f(\boldsymbol x) < t\)将因变量值(标签)判为正例或者反例。 对每个\(t\), 都可以在训练集上或者测试集上计算对应的假阳性率(健康人判入有病的比例)和真阳性率(病人判为有病的概率)。 \(t\)值取得越大, 相当于检验水平\(\alpha\)取值越小, 越不容易判为有病, 这时假阳性率低, 真阳性率也低; \(t\)值取得越小, 越容易判为有病, 相当于检验水平\(\alpha\)取值很大, 这时假阳性率很高,真阳性率也很高。 最好是对比较大的分界点\(t\), 当假阳性率很低时, 真阳性率就很高。 以给定\(t\)后的假阳性率为横坐标, 真阳性率为纵坐标作曲线, 称为ROC(Receiver Operating Curve,受试者工作特征)曲线(例子见46.2)。 曲线越靠近左上角越好。 可以用ROC曲线下方的面积大小度量判别方法的优劣, 面积越大越好。 记这个面积为AUC(Area Under the Curve)。
46.5.3.2 心脏病诊断示例
为了演示二分类问题, 使用Heart数据。 这是心脏病诊断的数据, 因变量AHD为是否有心脏病, 试图用各个自变量预测(分类)。
读入Heart数据集,并去掉有缺失值的观测:
Heart <- read_csv("data/Heart.csv") |>
select(-1) |>
mutate(
ChestPain = factor(ChestPain, levels = c(
"asymptomatic", "nonanginal",
"nontypical", "typical")),
Thal = factor(Thal, levels = c(
"fixed", "normal", "reversable")),
AHD = factor(AHD, levels = c("Yes", "No")) ) |>
drop_na()## New names:
## Rows: 303 Columns: 15
## ── Column specification
## ──────────────────────────────────────────────────────── Delimiter: "," chr
## (3): ChestPain, Thal, AHD dbl (12): ...1, Age, Sex, RestBP, Chol, Fbs, RestECG,
## MaxHR, ExAng, Oldpeak,...
## ℹ Use `spec()` to retrieve the full column specification for this data. ℹ
## Specify the column types or set `show_col_types = FALSE` to quiet this message.
## • `` -> `...1`## tibble [297 × 14] (S3: tbl_df/tbl/data.frame)
## $ Age : num [1:297] 63 67 67 37 41 56 62 57 63 53 ...
## $ Sex : num [1:297] 1 1 1 1 0 1 0 0 1 1 ...
## $ ChestPain: Factor w/ 4 levels "asymptomatic",..: 4 1 1 2 3 3 1 1 1 1 ...
## $ RestBP : num [1:297] 145 160 120 130 130 120 140 120 130 140 ...
## $ Chol : num [1:297] 233 286 229 250 204 236 268 354 254 203 ...
## $ Fbs : num [1:297] 1 0 0 0 0 0 0 0 0 1 ...
## $ RestECG : num [1:297] 2 2 2 0 2 0 2 0 2 2 ...
## $ MaxHR : num [1:297] 150 108 129 187 172 178 160 163 147 155 ...
## $ ExAng : num [1:297] 0 1 1 0 0 0 0 1 0 1 ...
## $ Oldpeak : num [1:297] 2.3 1.5 2.6 3.5 1.4 0.8 3.6 0.6 1.4 3.1 ...
## $ Slope : num [1:297] 3 2 2 3 1 1 3 1 2 3 ...
## $ Ca : num [1:297] 0 3 2 0 0 0 2 0 1 0 ...
## $ Thal : Factor w/ 3 levels "fixed","normal",..: 1 2 3 2 2 2 2 2 3 3 ...
## $ AHD : Factor w/ 2 levels "Yes","No": 2 1 1 2 2 2 1 2 1 1 ...产生训练集和测试集:
set.seed(101)
split <- initial_split(Heart, prop = 0.60)
heart_train <- training(split)
heart_test <- testing(split)预处理步骤、建立工作流程、在训练集上拟合模型:
heart_rec <-
recipe(AHD ~ .,
data = heart_train) |>
step_dummy(all_nominal_predictors())
lgs_model <- logistic_reg() |> set_engine("glm")
lgs_wflow <-
workflow() |>
add_model(lgs_model) |>
add_recipe(heart_rec)
lgs_fit <- fit(lgs_wflow, heart_train)在测试集上预测, 生成预测类别、每类的后验概率, 并添加真实类别结果:
heart_test_res <- predict(lgs_fit,
new_data = heart_test |> select(-AHD)) |>
bind_cols(predict(lgs_fit,
new_data = heart_test |> select(-AHD),
type = "prob") ) |>
bind_cols(heart_test |> select(AHD))
heart_test_res |>
slice(1:5) |>
knitr::kable()| .pred_class | .pred_Yes | .pred_No | AHD |
|---|---|---|---|
| No | 0.1756546 | 0.8243454 | No |
| No | 0.4230829 | 0.5769171 | No |
| No | 0.0205135 | 0.9794865 | No |
| No | 0.1867585 | 0.8132415 | No |
| No | 0.0125266 | 0.9874734 | No |
显示混淆矩阵:
## Truth
## Prediction Yes No
## Yes 32 4
## No 15 68注意混淆矩阵有两种做法, 这里是机器学习中常用的做法, 真实类别为两列, 预测类别为两行。 相当于如下命令的结果:
## AHD
## .pred_class Yes No
## Yes 32 4
## No 15 68另一种做法是转置显示。
计算准确率, 即预测类别等于真实类别的样例的比例:
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy binary 0.840Matthew相关系数:
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 mcc binary 0.665计算F1指标:
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 f_meas binary 0.771同时产生多个评估指标的示例:
classification_metrics <- metric_set(
accuracy, mcc, f_meas)
classification_metrics(heart_test_res,
truth = AHD,
estimate = .pred_class)## # A tibble: 3 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy binary 0.840
## 2 mcc binary 0.665
## 3 f_meas binary 0.771准确率和MCC同时评估两类的正确预测,
而F1指标更强调能够将正例检出。
tidymodels默认第一个水平为正例,
可以用event_level选项指定一个水平作为正例(事件)。
在这一点上R语言的不同函数有不同的规定。
ROC曲线、计算AUC值,
则需要使用预测的后验概率而不是类别。
用roc_curve()计算ROC曲线上点的坐标,如:
## Warning: Returning more (or less) than 1 row per `summarise()` group was deprecated in
## dplyr 1.1.0.
## ℹ Please use `reframe()` instead.
## ℹ When switching from `summarise()` to `reframe()`, remember that `reframe()`
## always returns an ungrouped data frame and adjust accordingly.
## ℹ The deprecated feature was likely used in the yardstick package.
## Please report the issue at <]8;;https://github.com/tidymodels/yardstick/issueshttps://github.com/tidymodels/yardstick/issues]8;;>.
## This warning is displayed once every 8 hours.
## Call `lifecycle::last_lifecycle_warnings()` to see where this warning was
## generated.| .threshold | specificity | sensitivity |
|---|---|---|
| -Inf | 0.0000000 | 1 |
| 0.0033245 | 0.0000000 | 1 |
| 0.0072132 | 0.0138889 | 1 |
| 0.0081136 | 0.0277778 | 1 |
| 0.0113795 | 0.0416667 | 1 |
用autoplot()从上面的数据集作AOC图:
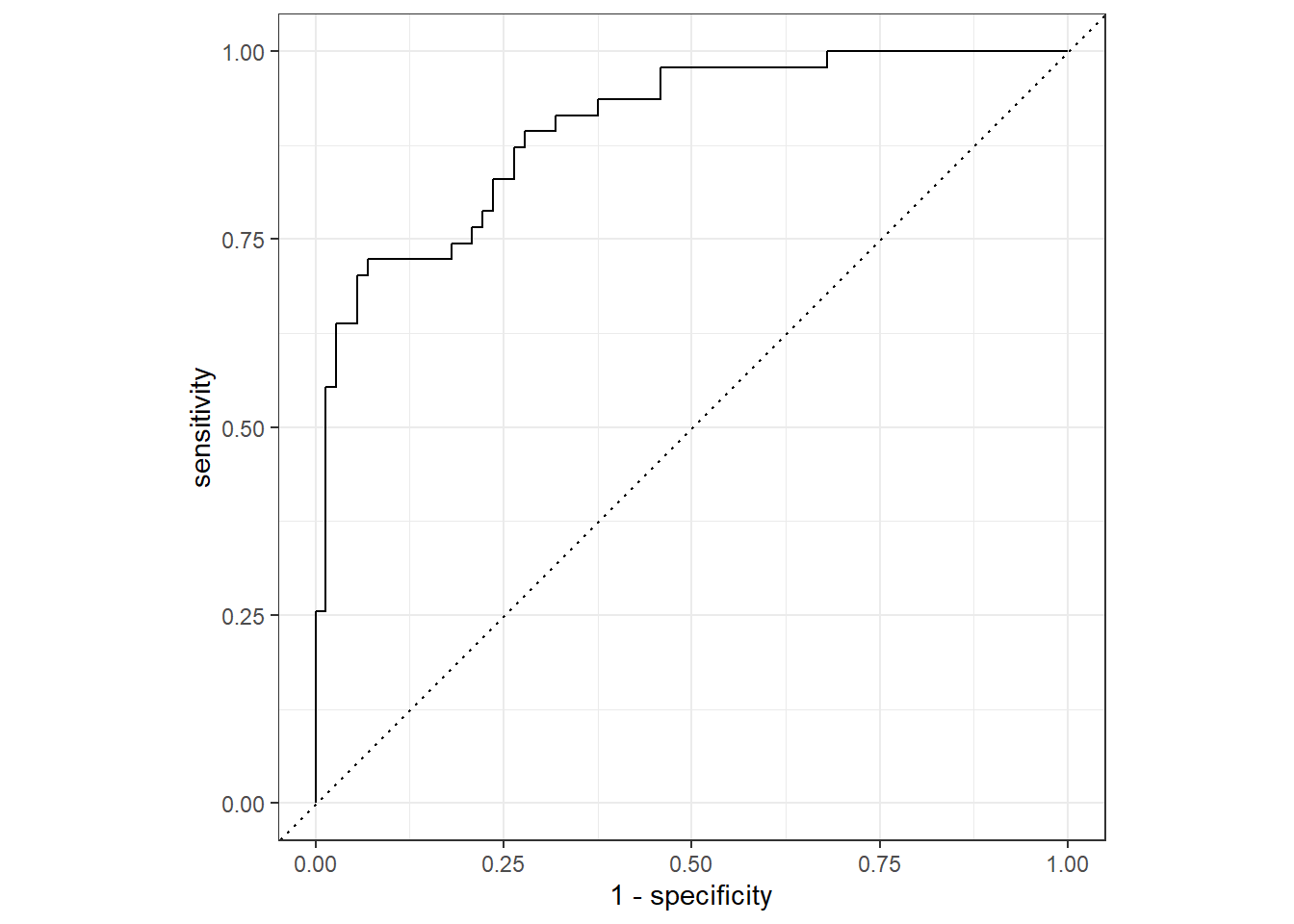
图46.2: 心脏病诊断建模ROC曲线
计算AUC值:
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 roc_auc binary 0.90046.5.4 多类别分类问题的评估
用鸢尾花数据作为示例。 这里不拆分训练集和测试集, 用交叉验证方法进行模型评估。 关于交叉验证方法, 见47.1.2.1。
用随机森林作判别, 建立工作流程并拟合:
data(iris)
iris_rec <-
recipe(Species ~ .,
data = iris)
rf_model <- rand_forest(
trees = 100,
min_n = 5 ) |>
set_engine("ranger") |>
set_mode("classification")
rf_wflow <-
workflow() |>
add_model(rf_model) |>
add_recipe(iris_rec)产生交叉验证分组, 用交叉验证样本拟合并评估:
set.seed(101)
iris_folds <- vfold_cv(iris, v = 10)
keep_pred <- control_resamples(
save_pred = TRUE, save_workflow = TRUE)
iris_cv <- rf_wflow |>
fit_resamples(resamples = iris_folds,
control = keep_pred)## Warning: package 'ranger' was built under R version 4.2.3从交叉验证结果中提取预测值、后验概率:
## tibble [150 × 8] (S3: tbl_df/tbl/data.frame)
## $ id : chr [1:150] "Fold01" "Fold01" "Fold01" "Fold01" ...
## $ .pred_setosa : num [1:150] 1 1 1 0 0 0.002 0 0.002 0 0 ...
## $ .pred_versicolor: num [1:150] 0 0 0 0.851 1 ...
## $ .pred_virginica : num [1:150] 0 0 0 0.149 0 ...
## $ .row : int [1:150] 5 9 46 53 83 90 93 99 116 124 ...
## $ .pred_class : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 2 2 2 2 2 3 3 ...
## $ Species : Factor w/ 3 levels "setosa","versicolor",..: 1 1 1 2 2 2 2 2 3 3 ...
## $ .config : chr [1:150] "Preprocessor1_Model1" "Preprocessor1_Model1" "Preprocessor1_Model1" "Preprocessor1_Model1" ...显示混淆矩阵:
## Truth
## Prediction setosa versicolor virginica
## setosa 50 0 0
## versicolor 0 47 3
## virginica 0 3 47准确率:
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 accuracy multiclass 0.96MCC指标也可以推广到多类别分类,如:
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 mcc multiclass 0.94原来适用于二分类的评价指标都可以设法推广到多类别分类问题, 推广方法有(设有\(k\)个类别):
- 宏平均法,将每个类别与“其它”类别作二分类计算, 将获得的\(k\)个评估指标计算平均值。
- 宏加权法,类似宏平均法, 但是计算平均值时按每类的样例数加权。
- 微平均法,对每个类分别计算对评估指标的贡献, 将这些贡献汇总计算成一个单一指标。
用estimator选项可以指定推广方法。
例如,
sensitivity()在二分类问题中计算敏感度(将正例识别为正例的概率),
用宏平均法推广到多分类:
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 sensitivity macro 0.96在上例中, 每一个类别都轮流作为正例, 然后将所得的3个敏感度平均。
宏加权法用estimator = "macro_weighted",
微平均法用estimator = "micro"。
计算AUC的推广指标,使用Hand and Till(2001)文章的方法:
## # A tibble: 1 × 3
## .metric .estimator .estimate
## <chr> <chr> <dbl>
## 1 roc_auc hand_till 0.996文献:
- Hand, D, and R Till. 2001. “A Simple Generalisation of the Area Under the ROC Curve for Multiple Class Classification Problems.” Machine Learning 45 (August): 171–86.