45 用H2O包进行统计学习
45.1 介绍
H2O是一个开源的、集成的机器学习环境, 基于Java语言开发, 支持并行处理, 支持大型数据。 R的H2O扩展包提供了对H2O软件的接口, 可以用比较统一的界面访问各种机器学习方法。
H2O使用自己的数据格式,
R的data.frame和data.table可以用as.h2o()函数转换为H2O的H2OFrame格式。
H2O的R扩展包利用网络服务访问正在运行的H2O软件, R本身并不进行计算和数据存储。
45.2 安装
如果安装有旧的H2O版本, 应预先卸载。 H2O包还依赖RCurl包和jsonlite包, 应提前安装。
H2O需要使用Java语言, 所以应该先安装一个Java环境, 64位JRE即可(Java运行环境), 64位的JDK则可以支持Java源代码编译和H2O测试。 在Windows下, 下载链接为:
H2O需要进行源代码编译, 所以在Windows操作系统中使用需要安装RTools工具包。
从下列链接下载H2O的源代码形式的扩展包:
放在当前工作目录后, 用如下命令安装, 安装时需要进行编译:
45.3 启动和退出H2O
启动:
library(h2o)
h2o.init(
nthreads = -1, max_mem_size = '16g',
ip = "127.0.0.1", port = 54321)
h2o.no_progress()因为启动了一个本地服务, 所以退出H2O时应该有一个关闭动作:
45.4 Hitters数据示例
转换数据格式为H2O格式:
拆分训练集、测试集:
splits <- h2o.splitFrame(
data = hf_hit,
ratios = c(0.60), seed = 1234)
train <- splits[[1]]
test <- splits[[2]]设置自变量、因变量:
用GBM方法。 先人为指定调优参数进行测试:
gbm1 <- h2o.gbm(
y=y, x=x,
training_frame = train,
ntrees = 10,
max_depth = 2,
min_rows = 3,
learn_rate = 0.1,
distribution= "gaussian")迭代过程的显示:
结果略。
训练集上的表现:
H2ORegressionMetrics: gbm
** Reported on training data. **
MSE: 98531.6
RMSE: 313.8974
MAE: 223.4093
RMSLE: 0.6764681
Mean Residual Deviance : 98531.6训练集上的RMSE为314。
变量重要度的度量:
Variable Importances:
variable relative_importance scaled_importance percentage
1 CRBI 34232028.000000 1.000000 0.288900
2 CHits 22870448.000000 0.668101 0.193014
3 Walks 21664550.000000 0.632874 0.182837
4 Runs 11754988.000000 0.343392 0.099206
5 CAtBat 8579465.000000 0.250627 0.072406
6 AtBat 5039900.000000 0.147228 0.042534
7 Hits 4386617.000000 0.128144 0.037021
8 CHmRun 3640191.250000 0.106339 0.030721
9 CRuns 3588647.750000 0.104833 0.030286
10 RBI 1586647.750000 0.046350 0.013390
11 CWalks 1147642.875000 0.033525 0.009685
12 HmRun 0.000000 0.000000 0.000000
13 Years 0.000000 0.000000 0.000000
14 League 0.000000 0.000000 0.000000
15 Division 0.000000 0.000000 0.000000
16 PutOuts 0.000000 0.000000 0.000000
17 Assists 0.000000 0.000000 0.000000
18 Errors 0.000000 0.000000 0.000000
19 NewLeague 0.000000 0.000000 0.000000可以用结果中scaled_importance作为每个变量重要程度的度量。
可以用条形图显示:
图形略。
下面进行参数调优。 H2O有两种参数调优方法, 第一种方法是将每个参数的若干个可能的值进行完全组合, 形成一个完全设计试验方案, 称为一个网格, 然后对每一种参数组合训练一个模型, 用交叉验证或者验证集比较这些模型; 第二种方法是形成了网格后, 在网格中随机均匀抽取进行模型比较, 这种方法可以设置一个时间限制, 在限制时间内找到较优模型, 其网格可以密集一些。
例如, 若参数\(A\)可取\(0.5, 1.5\), \(B\)可取\(10, 20\), \(C\)可取\(0.01, 0.1\), 则网格(完全试验方案)为: \[ \begin{array}{rlll} \text{NO} & A & B & C \\ 1 & 0.5 & 10 & 0.01 \\ 2 & 0.5 & 10 & 0.1 \\ 3 & 0.5 & 20 & 0.01 \\ 4 & 0.5 & 20 & 0.1 \\ 5 & 1.5 & 10 & 0.01 \\ 6 & 1.5 & 10 & 0.1 \\ 7 & 1.5 & 20 & 0.01 \\ 8 & 1.5 & 20 & 0.1 \end{array} \]
先用一个较小的网格搜索。 用默认的交叉验证方法。 仅修改树棵数、树最大深度、学习率参数。
time0 <- proc.time()[3]
gbm_params1 <- list(
ntrees = c(10, 20, 30),
max_depth = c(3, 5, 10),
min_rows = c(3, 5, 10),
learn_rate = c(0.01, 0.1, 0.5))
gbm_grid1 <- h2o.grid(
"gbm",
x = x,
y = y,
grid_id = "gbm_grid1",
training_frame = train,
nfolds=5,
seed = 1,
hyper_params= gbm_params1)
time_search <- paste(
round((proc.time()[3] - time0)/60), "minuntes")
cat("Time used:", time_search, "\n")
gbm_gridperf1 <- h2o.getGrid(
grid_id = "gbm_grid1",
sort_by = "rmse",
decreasing = FALSE)
gbm_gridperf1@summary_tableHyper-Parameter Search Summary: ordered by increasing rmse
learn_rate max_depth min_rows ntrees model_ids rmse
1 0.50000 10.00000 3.00000 10.00000 gbm_grid1_model_9 353.58941
2 0.50000 10.00000 3.00000 20.00000 gbm_grid1_model_36 355.47727
3 0.50000 10.00000 3.00000 30.00000 gbm_grid1_model_63 355.57809
4 0.10000 5.00000 3.00000 30.00000 gbm_grid1_model_59 356.84165
5 0.10000 10.00000 3.00000 30.00000 gbm_grid1_model_62 357.29143
---
learn_rate max_depth min_rows ntrees model_ids rmse
76 0.01000 3.00000 10.00000 10.00000 gbm_grid1_model_19 480.13855
77 0.01000 5.00000 3.00000 10.00000 gbm_grid1_model_4 480.30282
78 0.01000 5.00000 5.00000 10.00000 gbm_grid1_model_13 481.28910
79 0.01000 3.00000 5.00000 10.00000 gbm_grid1_model_10 481.49020
80 0.01000 10.00000 5.00000 10.00000 gbm_grid1_model_16 481.72192
81 0.01000 3.00000 3.00000 10.00000 gbm_grid1_model_1 481.72711完成参数网格优化后,
可以用h2o.getGrid()从优化结果中获取网格参数对应的各个模型,
并可以按RMSE、AOC等指标对模型排序显示。
可以用模型代码访问其中的具体模型。
最优参数组合为:
ntrees = 10;max_depth = 10;learn_rate = 0.5;min_rows = 3。
交叉验证的RMSE为354。
目前的最优模型:
此模型的变量重要度度量:
结果略, 与gbm1的排序有较大变化。
在最优组合附近再次进行搜索, 但使用离散随机化搜索策略, 取一个较密集的网格, 限制时间为5分钟:
time0 <- proc.time()[3]
gbm_params2 <- list(
ntrees = seq(5, 50, by=5),
max_depth = seq(1, 20, by=1),
min_rows = seq(2, 20, by=1),
learn_rate = c(0.01*(5:9), 0.1*(1:5)))
search_criteria2 <- list(
strategy = "RandomDiscrete",
max_runtime_secs = 300)
gbm_grid2 <- h2o.grid(
"gbm",
x = x,
y = y,
grid_id = "gbm_grid2",
training_frame = train,
nfolds = 5,
seed = 1,
hyper_params= gbm_params2,
search_criteria = search_criteria2)
time_search <- paste(
round((proc.time()[3] - time0)/60), "minuntes")
cat("Time used:", time_search, "\n")
gbm_gridperf2 <- h2o.getGrid(
grid_id = "gbm_grid2",
sort_by = "rmse",
decreasing = FALSE)
gbm_gridperf2@summary_tableHyper-Parameter Search Summary: ordered by increasing rmse
learn_rate max_depth min_rows ntrees model_ids rmse
1 0.10000 3.00000 2.00000 30.00000 gbm_grid2_model_820 344.00923
2 0.05000 4.00000 2.00000 35.00000 gbm_grid2_model_1151 344.84749
3 0.50000 9.00000 3.00000 5.00000 gbm_grid2_model_1033 344.85596
4 0.07000 15.00000 3.00000 50.00000 gbm_grid2_model_884 346.27286
5 0.07000 18.00000 3.00000 40.00000 gbm_grid2_model_675 347.23740
---
learn_rate max_depth min_rows ntrees model_ids rmse
1353 0.05000 18.00000 6.00000 5.00000 gbm_grid2_model_1197 455.81170
1354 0.05000 2.00000 2.00000 5.00000 gbm_grid2_model_1121 456.76030
1355 0.05000 2.00000 3.00000 5.00000 gbm_grid2_model_1305 458.97621
1356 0.08000 1.00000 5.00000 5.00000 gbm_grid2_model_571 459.82785
1357 0.06000 1.00000 5.00000 5.00000 gbm_grid2_model_793 469.72791
1358 0.05000 1.00000 2.00000 5.00000 gbm_grid2_model_167 473.67680最优参数组合:
ntrees = 30;max_depth = 3;learn_rate = 0.1;min_rows = 2。
交叉核实的RMSE为344。
提取调优结果的最优模型:
使用最后找到的最优模型在测试集上进行预测比较:
H2ORegressionMetrics: gbm
MSE: 77536.85
RMSE: 278.4544
MAE: 175.6084
RMSLE: 0.5015645
Mean Residual Deviance : 77536.85测试集上的RMSE为278, 比较理想。
变量重要度分析:
Variable Importances:
Variable Relative Importance Scaled Importance Percentage
1 CRBI 132.466204 1.000000 0.222229
2 Walks 96.518919 0.728631 0.161923
3 CHits 59.025072 0.445586 0.099022
4 CHmRun 55.486735 0.418875 0.093086
5 Runs 50.785759 0.383387 0.085200
6 CRuns 38.727044 0.292354 0.064970
7 CAtBat 33.082905 0.249746 0.055501
8 Hits 25.749126 0.194383 0.043197
9 CWalks 20.566305 0.155257 0.034503
10 RBI 16.308211 0.123112 0.027359
11 Years 15.404481 0.116290 0.025843
12 AtBat 14.144269 0.106776 0.023729
13 Errors 14.052819 0.106086 0.023575
14 PutOuts 10.156549 0.076673 0.017039
15 HmRun 5.738121 0.043318 0.009626
16 Division 4.937515 0.037274 0.008283
17 NewLeague 1.975050 0.014910 0.003313
18 Assists 0.954782 0.007208 0.001602重要度作图:
图形略。
在测试集上计算因变量预测值:
predict
1 423.0851
2 890.0124
3 160.4647
4 815.4056
5 1271.8789
6 181.5200变量解释性分析:
这会产生多个关于每个变量的贡献的图形。 也有一些单个图形的函数, 比如SHAP概况图:
SHAP计算每个观测上每个变量的贡献值, 并对变量的总的贡献由大到小排序, 并用散点图绘制出这些贡献。 结果如:
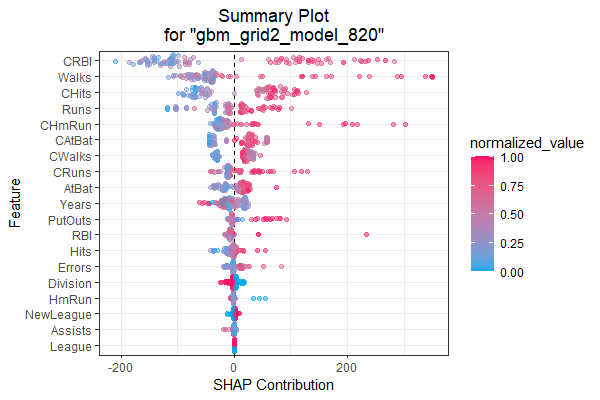
变量重要度图:

45.5 AutoML
H2O提供了一个AutoML功能, 可以自动使用各个机器学习方法进行训练、参数调优、模型比较, 输出占优的多个模型。
用户仅需要指定训练数据集training_frame、因变量y、最多允许训练时间max_runtime_secs,
自变量自动选择为因变量以外的所有变量,
参数调优自动使用交叉验证方法。
示例:
library(h2o)
h2o.init()
train <- h2o.importFile("https://s3.amazonaws.com/erin-data/higgs/higgs_train_10k.csv")
test <- h2o.importFile("https://s3.amazonaws.com/erin-data/higgs/higgs_test_5k.csv")
y <- "response"
x <- setdiff(names(train), y)
# 分类问题的因变量必须是因子
train[, y] <- as.factor(train[, y])
test[, y] <- as.factor(test[, y])
# 限制5分钟
aml <- h2o.automl(
x = x, y = y,
training_frame = train,
#max_models = 20,
max_runtime_secs = 300,
seed = 1)
# View the AutoML Leaderboard
lb <- aml@leaderboard
print(lb, n = nrow(lb)) model_id auc logloss aucpr
1 StackedEnsemble_AllModels_3_AutoML_1_20230717_82125 0.7896537 0.5492908 0.8084317
2 StackedEnsemble_AllModels_4_AutoML_1_20230717_82125 0.7888052 0.5503257 0.8076245
3 StackedEnsemble_AllModels_2_AutoML_1_20230717_82125 0.7874863 0.5515817 0.8072801
4 StackedEnsemble_AllModels_1_AutoML_1_20230717_82125 0.7867515 0.5522508 0.8069401
5 StackedEnsemble_BestOfFamily_4_AutoML_1_20230717_82125 0.7854556 0.5534061 0.8053178
6 StackedEnsemble_BestOfFamily_5_AutoML_1_20230717_82125 0.7847936 0.5542375 0.8050583
7 StackedEnsemble_BestOfFamily_3_AutoML_1_20230717_82125 0.7832484 0.5556922 0.8029427
8 StackedEnsemble_BestOfFamily_2_AutoML_1_20230717_82125 0.7819484 0.5568627 0.8017783
9 StackedEnsemble_AllModels_5_AutoML_1_20230717_82125 0.7817324 0.5638011 0.7997335
10 StackedEnsemble_BestOfFamily_1_AutoML_1_20230717_82125 0.7800970 0.5592433 0.7990314
11 GBM_grid_1_AutoML_1_20230717_82125_model_12 0.7800394 0.5595114 0.8014000
12 GBM_grid_1_AutoML_1_20230717_82125_model_9 0.7797381 0.5625036 0.7983718
13 GBM_1_AutoML_1_20230717_82125 0.7795121 0.5602557 0.7995356
14 GBM_2_AutoML_1_20230717_82125 0.7792939 0.5608256 0.7984392
15 GBM_grid_1_AutoML_1_20230717_82125_model_17 0.7790189 0.5649027 0.7959446
16 GBM_grid_1_AutoML_1_20230717_82125_model_16 0.7788996 0.5624376 0.7947606
17 GBM_5_AutoML_1_20230717_82125 0.7788048 0.5617556 0.7967867
18 GBM_grid_1_AutoML_1_20230717_82125_model_19 0.7786671 0.5639216 0.7971413
19 StackedEnsemble_BestOfFamily_6_AutoML_1_20230717_82125 0.7779028 0.5602988 0.7989296
20 GBM_grid_1_AutoML_1_20230717_82125_model_2 0.7778602 0.5646552 0.7953585
21 GBM_grid_1_AutoML_1_20230717_82125_model_14 0.7775555 0.5668371 0.7924693
22 GBM_grid_1_AutoML_1_20230717_82125_model_6 0.7772192 0.5642876 0.7954070
23 GBM_grid_1_AutoML_1_20230717_82125_model_7 0.7764426 0.5701478 0.7923477
24 GBM_3_AutoML_1_20230717_82125 0.7751876 0.5650460 0.7946101
25 GBM_4_AutoML_1_20230717_82125 0.7742870 0.5656442 0.7963992
26 GBM_grid_1_AutoML_1_20230717_82125_model_11 0.7734054 0.5716275 0.7919521
27 GBM_grid_1_AutoML_1_20230717_82125_model_3 0.7729262 0.5681808 0.7911955
28 GBM_grid_1_AutoML_1_20230717_82125_model_4 0.7705223 0.5692442 0.7890998
29 GBM_grid_1_AutoML_1_20230717_82125_model_5 0.7704555 0.5732127 0.7881083
30 XRT_1_AutoML_1_20230717_82125 0.7642216 0.5814393 0.7820797
31 DRF_1_AutoML_1_20230717_82125 0.7631956 0.5802385 0.7840833
32 GBM_grid_1_AutoML_1_20230717_82125_model_10 0.7603439 0.5805147 0.7762872
33 GBM_grid_1_AutoML_1_20230717_82125_model_8 0.7532375 0.5947734 0.7703927
34 GBM_grid_1_AutoML_1_20230717_82125_model_15 0.7532095 0.5887163 0.7719831
35 GBM_grid_1_AutoML_1_20230717_82125_model_1 0.7476579 0.5915102 0.7632106
36 GBM_grid_1_AutoML_1_20230717_82125_model_13 0.7426757 0.6044879 0.7619594
37 DeepLearning_grid_2_AutoML_1_20230717_82125_model_1 0.7297311 0.6137454 0.7358833
38 DeepLearning_grid_1_AutoML_1_20230717_82125_model_1 0.7265855 0.6634738 0.7275126
39 GBM_grid_1_AutoML_1_20230717_82125_model_18 0.7245035 0.6152842 0.7447474
40 DeepLearning_grid_3_AutoML_1_20230717_82125_model_1 0.7160532 0.6232399 0.7192921
41 DeepLearning_grid_1_AutoML_1_20230717_82125_model_2 0.7142102 0.6319313 0.7162592
42 DeepLearning_1_AutoML_1_20230717_82125 0.7081655 0.6274959 0.7123640
43 DeepLearning_grid_1_AutoML_1_20230717_82125_model_3 0.7042074 0.6544330 0.7070402
44 GLM_1_AutoML_1_20230717_82125 0.6826483 0.6385202 0.6807189
mean_per_class_error rmse mse
1 0.3281307 0.4317858 0.1864389
2 0.3212199 0.4322351 0.1868272
3 0.3315550 0.4328426 0.1873527
4 0.3280114 0.4331952 0.1876580
5 0.3354530 0.4338531 0.1882285
6 0.3293683 0.4341424 0.1884796
7 0.3363375 0.4347748 0.1890291
8 0.3316707 0.4353819 0.1895574
9 0.3214789 0.4371155 0.1910699
10 0.3486560 0.4362937 0.1903522
11 0.3367738 0.4365091 0.1905402
12 0.3330875 0.4371565 0.1911058
13 0.3275111 0.4366086 0.1906271
14 0.3278906 0.4367848 0.1907810
15 0.3285993 0.4379804 0.1918268
16 0.3347428 0.4372481 0.1911859
17 0.3343263 0.4371076 0.1910630
18 0.3363475 0.4377541 0.1916287
19 0.3301687 0.4370242 0.1909902
20 0.3337600 0.4380880 0.1919211
21 0.3236620 0.4387509 0.1925023
22 0.3248413 0.4380742 0.1919090
23 0.3332508 0.4402129 0.1937874
24 0.3302285 0.4388332 0.1925746
25 0.3456632 0.4393214 0.1930033
26 0.3288446 0.4411005 0.1945696
27 0.3228082 0.4399974 0.1935977
28 0.3497369 0.4407917 0.1942973
29 0.3286788 0.4424870 0.1957948
30 0.3474700 0.4457808 0.1987205
31 0.3492529 0.4455428 0.1985084
32 0.3560809 0.4456789 0.1986297
33 0.3445959 0.4515736 0.2039187
34 0.3537379 0.4498069 0.2023263
35 0.3594190 0.4510526 0.2034484
36 0.3540427 0.4563462 0.2082518
37 0.3674737 0.4596807 0.2113064
38 0.3713030 0.4701575 0.2210480
39 0.3957685 0.4618223 0.2132799
40 0.3822944 0.4643025 0.2155768
41 0.3894767 0.4655305 0.2167187
42 0.3786903 0.4666819 0.2177920
43 0.4008949 0.4726119 0.2233620
44 0.3972341 0.4726827 0.2234289