24 数据汇总
24.1 用dplyr作数据汇总
dplyr包的summarise()函数可以对数据框计算统计量。
以肺癌病人化疗数据cancer.csv为例,
有34个肺癌病人的数据:
## Rows: 34 Columns: 6
## ── Column specification ────────────────────────────────────────────────────────
## Delimiter: ","
## chr (2): sex, type
## dbl (4): id, age, v0, v1
##
## ℹ Use `spec()` to retrieve the full column specification for this data.
## ℹ Specify the column types or set `show_col_types = FALSE` to quiet this message.| id | age | sex | type | v0 | v1 |
|---|---|---|---|---|---|
| 1 | 70 | F | 腺癌 | 26.51 | 2.91 |
| 2 | 70 | F | 腺癌 | 135.48 | 35.08 |
| 3 | 69 | F | 腺癌 | 209.74 | 74.44 |
| 4 | 68 | M | 腺癌 | 61.00 | 34.97 |
| 5 | 67 | M | 鳞癌 | 237.75 | 128.34 |
| 6 | 75 | F | 腺癌 | 330.24 | 112.34 |
| 7 | 52 | M | 鳞癌 | 104.90 | 32.10 |
| 8 | 71 | M | 鳞癌 | 85.15 | 29.15 |
| 9 | 68 | M | 鳞癌 | 101.65 | 22.15 |
| 10 | 79 | M | 鳞癌 | 65.54 | 21.94 |
| 11 | 55 | M | 腺癌 | 125.31 | 12.33 |
| 12 | 54 | M | 鳞癌 | 224.36 | 99.44 |
| 13 | 55 | F | 腺癌 | 12.93 | 2.30 |
| 14 | 75 | M | 腺癌 | 40.21 | 23.96 |
| 15 | 61 | F | 腺癌 | 12.58 | 7.39 |
| 16 | 76 | M | 鳞癌 | 231.04 | 112.58 |
| 17 | 65 | M | 鳞癌 | 172.13 | 91.62 |
| 18 | 66 | M | 鳞癌 | 39.26 | 13.95 |
| 19 | NA | F | 腺癌 | 32.91 | 9.45 |
| 20 | 63 | F | 腺癌 | 161.00 | 122.45 |
| 21 | 67 | M | 鳞癌 | 105.26 | 68.35 |
| 22 | 51 | M | 鳞癌 | 13.25 | 5.25 |
| 23 | 49 | M | 鳞癌 | 18.70 | 3.34 |
| 24 | 49 | M | 鳞癌 | 60.23 | 50.36 |
| 25 | NA | F | 鳞癌 | 223.00 | 25.59 |
| 26 | NA | M | 鳞癌 | 145.70 | 35.36 |
| 27 | NA | M | 鳞癌 | 138.44 | 11.30 |
| 28 | NA | M | 鳞癌 | 83.71 | 76.45 |
| 29 | NA | M | 鳞癌 | 74.48 | 23.66 |
| 30 | NA | F | 腺癌 | 42.70 | 5.97 |
| 31 | NA | M | 鳞癌 | 142.48 | 68.46 |
| 32 | NA | F | 鳞癌 | 46.97 | 27.32 |
| 33 | NA | F | 鳞癌 | 170.63 | 74.76 |
| 34 | NA | F | 鳞癌 | 67.37 | 54.52 |
求年龄(age)的观测个数、非缺失观测个数、平均值、标准差:
d.cancer |>
summarise(
nobs = n(),
n = sum(!is.na(age)),
mean.age=mean(age, na.rm=TRUE),
sd.age=sd(age, na.rm=TRUE)) |>
knitr::kable()| nobs | n | mean.age | sd.age |
|---|---|---|---|
| 34 | 23 | 64.13043 | 9.161701 |
其中n()计算观测数(行数)。
为了计算某列的非缺失值个数,用sum(!is.na(x))。
summarise中常用的汇总函数有:
- 位置度量:
mean(),median()。 - 分散程度(变异性)度量:
sd(),IQR(),mad()。 - 分位数:
min(),max(),quantile()。 - 按下标查询,如
first(x)取出x[1],last(x)取出x的最后一个元素,nth(x,2)取出x[2]。 可以提供一个缺省值以防某个下标位置不存在。 - 计数:
n()给出某个组的观测数,sum(!is.na(x))统计x的非缺失值个数,n_distinct(x)统计x的不同值个数(缺失值也算一个值), 也可以计算多个变量的不同组合值个数。 count(x)给出x的每个不同值的个数(类似于table()函数), 但count(x)只能单独作为管道运算的一个步骤, 不能用在summarise函数中。
这里有些函数是dplyr包提供的, 仅适用于tibble类型。
count()函数允许用wt添加一个权重,
实际是重复观测数。
例如,
有如下的汇总数据:
| age | n | meanheight |
|---|---|---|
| 10 | 20 | 110 |
| 10 | 10 | 112 |
| 11 | 25 | 120 |
其中n表示该观测代表的人数。
汇总两个年龄的人数:
## # A tibble: 2 × 2
## age n
## <dbl> <dbl>
## 1 10 30
## 2 11 25计算所有人的总数和平均身高:
## # A tibble: 1 × 2
## nobs meanheight
## <dbl> <dbl>
## 1 55 115.基本R和dplyr没有提供对加权计算的一般支持。
24.2 多个变量的汇总
在有多个变量需要汇总时,
summarise的格式就会比较罗嗦。
比如,
需要对cancer数据集中v0和v1两个变量同时计算平均值和标准差:
d.cancer |>
summarise(
mean_v0=mean(v0, na.rm=TRUE),
sd_v0 = sd(v0, na.rm=TRUE),
mean_1=mean(v1, na.rm=TRUE),
sd_v1 = sd(v1, na.rm=TRUE)) |>
knitr::kable(digits=2)| mean_v0 | sd_v0 | mean_1 | sd_v1 |
|---|---|---|---|
| 110.08 | 79.52 | 44.69 | 38.44 |
显然,如果有许多变量要计算不止一个统计量,
就需要人为地将每一个变量的每一个统计量单独命名。
dplyr包的across()函数可以指定一批变量名与一批统计函数,
自动命名结果变量。
如果仅计算一个统计量,
只要在across中给出要分析的变量集合与一个统计函数名,
如:
| v0 | v1 | age |
|---|---|---|
| 110.08 | 44.69 | NA |
其中age的平均值得到了缺失值。
在指定函数时,可以用R的无名函数方法,
增加选项:
d.cancer |>
summarise(
across(
c("v0", "v1", "age"),
\(x) mean(x, na.rm=TRUE))) |>
knitr::kable(digits=2)| v0 | v1 | age |
|---|---|---|
| 110.08 | 44.69 | 64.13 |
可以看出,
这样仅指定函数名或无名函数的方法,
仅限于一种统计量,
而且统计量结果名称也沿用了变量名本身。
调用across较好的方法,
是将要计算的统计量写在一个有名列表中,
列表的元素名将出现在结果的变量名中作为统计量名部分,
列表的元素是函数名或无名函数。
如:
d.cancer |>
summarise(
across(c("v0", "v1", "age"),
list(
avg = \(x) mean(x, na.rm=TRUE),
std = \(x) sd(x, na.rm=TRUE)))) |>
knitr::kable(digits=2)| v0_avg | v0_std | v1_avg | v1_std | age_avg | age_std |
|---|---|---|---|---|---|
| 110.08 | 79.52 | 44.69 | 38.44 | 64.13 | 9.16 |
across()的第一自变量是要分析的变量集合,
可以使用dplyr::select()的变量格式,
如v0, c(v0, v1), c("v0", "v1"),
starts_with("v"), 5:6等。
across()的第二自变量:
- 可以是一个函数,如
mean; - 可以是一个如
\(x) mean(x, na.rm=TRUE)这样的自定义无名函数; - 函数或者自定义无名函数的有名列表,
可以计算所有变量的所有统计函数值并对结果自动命名,
结果统计量自动命名为“
变量名_统计量名”的格式。
如果要对所有数值型变量计算某些统计量, 可以用
这样的格式。 如:
d.cancer |>
summarise(
across(where(is.numeric),
list(avg = \(x) mean(x, na.rm=TRUE),
std = \(x) sd(x, na.rm=TRUE)))) |>
knitr::kable(digits=2)| id_avg | id_std | age_avg | age_std | v0_avg | v0_std | v1_avg | v1_std |
|---|---|---|---|---|---|---|---|
| 17.5 | 9.96 | 64.13 | 9.16 | 110.08 | 79.52 | 44.69 | 38.44 |
其中的is.numeric用来筛选需要进行统计的变量子集,
可以替换成其它的示性函数(对每列返回一个逻辑值的函数),
如is.character, is.Date, is.POSIXct, is.logical,
is.factor。
为了对所有列都计算相同的统计量,
可以在across()中指定变量集合为everything(),如
d.cancer |>
select(v0, v1) |>
summarise(across(everything(),
list(avg = \(x) mean(x, na.rm=TRUE),
std = \(x) sd(x, na.rm=TRUE))) ) |>
knitr::kable(digits=2)| v0_avg | v0_std | v1_avg | v1_std |
|---|---|---|---|
| 110.08 | 79.52 | 44.69 | 38.44 |
可以用tidyr包将上面的变量与统计量拆开, 将变量放到不同的观测中,如:
d.cancer |>
select(v0, v1) |>
summarise(across(everything(),
list(avg = \(x) mean(x, na.rm=TRUE),
std = \(x) sd(x, na.rm=TRUE))) ) |>
pivot_longer(
everything(),
names_sep ="_",
names_to = c("variable", ".value")
) |>
knitr::kable(digits=2)| variable | avg | std |
|---|---|---|
| v0 | 110.08 | 79.52 |
| v1 | 44.69 | 38.44 |
24.2.1 修改多个变量
除了对多个变量进行汇总以外,
有时需要对多个变量进行修改。
例如,某个数据集中所有的数值型变量都用-999表示缺失值,
需要将其改为NA,
如果在mutate中逐个写出修改,
就太麻烦,
这时可以在mutate()中用across()指定要修改的列和修改方法,如:
d.999.orig <- tribble(
~id, ~a, ~b, ~c,
1, 5, 10, 6,
2, -999, 12, 8,
3, 4, -999, -999,
4, 7, 9, 10
)
d.999.mod <- d.999.orig |>
mutate(across(
a:c,
\(x) ifelse(x == -999, NA, x)
))
d.999.mod## # A tibble: 4 × 4
## id a b c
## <dbl> <dbl> <dbl> <dbl>
## 1 1 5 10 6
## 2 2 NA 12 8
## 3 3 4 NA NA
## 4 4 7 9 10修改结果直接替换了原来的变量。
如果希望修改结果用新变量保存,
可以在across中用.names指定一个新变量命名模式,
如
d.999 <- d.999.orig |>
mutate(across(
a:c,
\(x) ifelse(x == -999, NA, x),
.names = "{.col}_na"
))
d.999## # A tibble: 4 × 7
## id a b c a_na b_na c_na
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 5 10 6 5 10 6
## 2 2 -999 12 8 NA 12 8
## 3 3 4 -999 -999 4 NA NA
## 4 4 7 9 10 7 9 10其中.names的取值是一个模式字符串,
模式中的{.col}特指原始变量名。
下面的函数expand_dates(),
输入一个数据框,
选择其中的所有日期列,
将其每一个都拆分为年、月、日:
##
## Attaching package: 'lubridate'## The following objects are masked from 'package:base':
##
## date, intersect, setdiff, unionexpand_dates <- function(df){
df |>
mutate(across(
where(is.Date),
list(year = year, month = month, day = mday)
))
}d.dates <- read_csv(
'data/dates.csv',
locale=locale(encoding="GBK"),
col_types=cols(
`序号`=col_integer(),
`出生日期`=col_date(format="%Y/%m/%d"),
`发病日期`=col_date(format="%Y/%m/%d")
))
knitr::kable(d.dates[1:4,])| 序号 | 出生日期 | 发病日期 |
|---|---|---|
| 1 | 1941-03-08 | 2007-01-01 |
| 2 | 1972-01-24 | 2007-01-01 |
| 3 | 1932-06-01 | 2007-01-01 |
| 4 | 1947-05-17 | 2007-01-01 |
| 序号 | 出生日期 | 发病日期 | 出生日期_year | 出生日期_month | 出生日期_day | 发病日期_year | 发病日期_month | 发病日期_day |
|---|---|---|---|---|---|---|---|---|
| 1 | 1941-03-08 | 2007-01-01 | 1941 | 3 | 8 | 2007 | 1 | 1 |
| 2 | 1972-01-24 | 2007-01-01 | 1972 | 1 | 24 | 2007 | 1 | 1 |
| 3 | 1932-06-01 | 2007-01-01 | 1932 | 6 | 1 | 2007 | 1 | 1 |
| 4 | 1947-05-17 | 2007-01-01 | 1947 | 5 | 17 | 2007 | 1 | 1 |
24.2.2 针对多个变量进行筛选
across的类似函数也可以用在filter函数中,
同时指定多个变量进行筛选。
if_any指定是任一变量满足条件就选择该观测,
if_all指定所有变量满足该该条件才选择该观测。
如:
## # A tibble: 2 × 7
## id a b c a_na b_na c_na
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 2 -999 12 8 NA 12 8
## 2 3 4 -999 -999 4 NA NA## # A tibble: 2 × 7
## id a b c a_na b_na c_na
## <dbl> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
## 1 1 5 10 6 5 10 6
## 2 4 7 9 10 7 9 1024.3 用dplyr作数据分组汇总
数据汇总问题更常见的是作分组汇总。
dplyr包的group_by()函数对数据框(或tibble)分组,
随后的summarise()将按照分组汇总。
group_by()对数据框增加分组信息,
使得后续的管道操作针对每一组内进行操作,
但group_by()并不修改数据框内容。
24.3.1 简单汇总
比如, 按不同性别分组计算人数与年龄平均值:
d.cancer |>
group_by(sex) |>
summarise(
count = n(),
mean.age = mean(age, na.rm=TRUE)) |>
knitr::kable(digits=2)| sex | count | mean.age |
|---|---|---|
| F | 13 | 66.14 |
| M | 21 | 63.25 |
按不同性别分组计算v0和v1的人数、平均值、标准差:
d.cancer |>
group_by(sex) |>
summarise(across(
c("v0", "v1"),
list(
count = ~n(),
avg = \(x) mean(x, na.rm=TRUE),
std = \(x) sd(x, na.rm=TRUE))) ) |>
knitr::kable(digits=2)| sex | v0_count | v0_avg | v0_std | v1_count | v1_avg | v1_std |
|---|---|---|---|---|---|---|
| F | 13 | 113.24 | 100.07 | 13 | 42.66 | 41.72 |
| M | 21 | 108.12 | 66.45 | 21 | 45.96 | 37.28 |
上面的程序应该将v0_count和v1_count合并,
但是summarise在有多个变量和多个统计量时总会将变量两两搭配,
确实需要时可以分别统计再合并,结果程序会略繁琐一些:
bind_cols(
d.cancer |>
group_by(sex) |>
summarise(count = n() ),
d.cancer |>
group_by(sex) |>
summarise(
across(c("v0", "v1"),
list(
avg = \(x) mean(x, na.rm=TRUE),
std = \(x) sd(x, na.rm=TRUE)))) |>
select(-sex)
) |>
knitr::kable(digits=2)| sex | count | v0_avg | v0_std | v1_avg | v1_std |
|---|---|---|---|---|---|
| F | 13 | 113.24 | 100.07 | 42.66 | 41.72 |
| M | 21 | 108.12 | 66.45 | 45.96 | 37.28 |
24.3.2 满足条件汇总
为了查询数值型变量取值满足某种条件的个数和比例,
可以将该条件用sum()和mean()函数统计,
比如,
对男女两组分别计算年龄在60岁以上的人数和比例:
d.cancer |>
group_by(sex) |>
summarise(
nold = sum(age >= 60, na.rm=TRUE),
pold = nold / n()) |>
knitr::kable(digits=4)| sex | nold | pold |
|---|---|---|
| F | 6 | 0.4615 |
| M | 10 | 0.4762 |
24.3.3 分组筛选
用group_by()分组后除了可以分组汇总,
还可以分组筛选:
d.cancer |>
group_by(sex) |>
filter(rank(desc(v0)) <= 2) |>
arrange(sex, desc(v0)) |>
knitr::kable()| id | age | sex | type | v0 | v1 |
|---|---|---|---|---|---|
| 6 | 75 | F | 腺癌 | 330.24 | 112.34 |
| 25 | NA | F | 鳞癌 | 223.00 | 25.59 |
| 5 | 67 | M | 鳞癌 | 237.75 | 128.34 |
| 16 | 76 | M | 鳞癌 | 231.04 | 112.58 |
以上程序按性别分组后, 在每组中找出疗前体积排名在前两名的。
24.4 交叉分类的汇总
24.4.1 简单例子
下面的程序对d.cancer数据框分性别与病理类型分别统计人数:
## `summarise()` has grouped output by 'sex'. You can override using the `.groups`
## argument.| sex | type | freq |
|---|---|---|
| F | 鳞癌 | 4 |
| F | 腺癌 | 9 |
| M | 鳞癌 | 18 |
| M | 腺癌 | 3 |
如果仅需要计算交叉分类频数,
不需要用group_by(),
可以用dplyr的count()函数,如:
| sex | type | n |
|---|---|---|
| F | 鳞癌 | 4 |
| F | 腺癌 | 9 |
| M | 鳞癌 | 18 |
| M | 腺癌 | 3 |
下面的程序数出NHANES数据框中ID(受访者编码)与SurveyYr(考察年份)每一对组合的出现次数, 筛选出二次及以上者,并降序排列,仅显示前10行结果:
NHANES |>
count(ID, SurveyYr) |>
filter(n >= 2 ) |>
arrange(desc(n)) |>
slice_head(n = 10) |>
knitr::kable()| ID | SurveyYr | n |
|---|---|---|
| 70324 | 2011_12 | 8 |
| 62927 | 2011_12 | 7 |
| 63297 | 2011_12 | 7 |
| 69626 | 2011_12 | 7 |
| 60566 | 2009_10 | 6 |
| 61442 | 2009_10 | 6 |
| 63163 | 2011_12 | 6 |
| 63330 | 2011_12 | 6 |
| 63390 | 2011_12 | 6 |
| 63744 | 2011_12 | 6 |
24.4.2 分组信息残留问题
用group_by()交叉分组汇总后的结果不是普通的tibble,
总是带有外层分组信息,
最内层的分组信息不再有效。
如果有更多的分组变量,
每次用summarise()汇总都会去掉最内层的分组信息,
但保留其它层的分组信息。
不注意这种规定在后续的使用中可能会产生问题,
为此,
可以在summarise()函数中加选项.groups="drop"要求汇总后不保留任何分组信息,
或者汇总后再用ungroup()函数取消所有分组。
例如,
希望在用group_by()按照性别和病理类别交叉分类计算频数后求所有病人的总人数,
用了如下程序:
d.cancer |>
group_by(sex, type) |>
summarise(freq=n()) |>
summarise(ntotal=sum(freq)) |>
knitr::kable()## `summarise()` has grouped output by 'sex'. You can override using the `.groups`
## argument.| sex | ntotal |
|---|---|
| F | 13 |
| M | 21 |
可以看出并没有能够计算总人数,
而是按原来交叉分类的外层分类即性别分组计算了总人数。
这是因为交叉分组计算频数后的结果仍按照外层分类变量sex分组,
所以summarise(ntotal=sum(freq))也是分两组进行的。
在第一个summarise()中加.groups = "drop"可以不保留分组信息,如:
d.cancer |>
group_by(sex, type) |>
summarise(freq=n(), .groups="drop") |>
summarise(ntotal=sum(freq)) |>
knitr::kable()| ntotal |
|---|
| 34 |
得到了需要的结果。
为了避免在汇总后仍保留分组信息,
在summarise()中加选项.groups="drop",
这样可以在汇总结果中去掉残留的分组信息。
默认的选项是.groups="drop_last",
即仅去掉最内层分组信息。
还可以要求.groups="keep",
这时汇总结果中包含所有的分组信息,
包括最内层分组。
用ungroup()去掉所有分组信息的例子如下:
d.cancer |>
group_by(sex, type) |>
summarise(freq=n()) |>
ungroup() |>
summarise(ntotal=sum(freq)) |>
knitr::kable()## `summarise()` has grouped output by 'sex'. You can override using the `.groups`
## argument.| ntotal |
|---|
| 34 |
24.5 在自定义函数中使用dplyr功能
如果对数据框有一些通用的操作, 可以编写函数, 以数据框为输入和输出, 指定针对的变量集合与要进行的操作。
因为在mutate和summarise中需要使用直接写的变量名,
以字符型变量形式输入的变量名无法直接使用。
实际上,
dplyr和tidyr关于变量和变量集合使用两种写法类似、底层不同的做法,
即数据掩码(data masking)和整洁选择(tidy select)。
mutate(), filter(),
arrange(), count(),
group_by(), summarise()在底层使用数据掩码方法。
select(), relocate(),
rename(), pull(), across() 在底层使用整洁选择方法。
在使用数据掩码方法的函数中,
如果针对的数据框变量是原样(不是字符串或者字符型变量)输入的,
可以用{{ var_names }}的写法表示。
比如,
输入一个分组变量和一个分析变量,
计算均值和最大值:
group_summ_1 <- function(df, group_var, ana_var){
df |>
group_by({{ group_var }}) |>
summarise(
mean = mean({{ana_var}}, na.rm=TRUE),
max = max({{ana_var}}, na.rm=TRUE))
}
group_summ_1(d.class, sex, height)## # A tibble: 2 × 3
## sex mean max
## <fct> <dbl> <dbl>
## 1 M 63.9 72
## 2 F 60.6 66.5上面的group_by和summarise都是数据掩码方式指定变量的,
所以当调用自定义函数时如果原样写分组变量和分析变量名,
而不是写成字符串或者输入为字符型变量,
就只要用{{ var_name }}这种格式。
但是,如果输入为字符串则不行:
group_summ_1(d.class, "sex", "height")
## # A tibble: 1 × 3
## `"sex"` mean max
## <chr> <dbl> <chr>
## 1 sex NA height这时,
可以用.data[[var_name]]这种方式访问,如:
group_summ_2 <- function(df, group_var, ana_var){
df |>
group_by(.data[[group_var]]) |>
summarise(
mean = mean(.data[[ana_var]], na.rm=TRUE),
max = max(.data[[ana_var]], na.rm=TRUE))
}
group_summ_2(d.class, "sex", "height")## # A tibble: 2 × 3
## sex mean max
## <fct> <dbl> <dbl>
## 1 M 63.9 72
## 2 F 60.6 66.5基于数据掩码的函数在指定多个变量时,
为了能够使用整洁选择时可用的方法,
需要用pick()函数进行说明。
比如,
group_by()是基于数据掩码的,
如果要指定多个变量,
无法直接使用如c(sex, age)这样的方法,
可以在自定义函数中写成pick({{ group_var }}),
如:
group_summ_3 <- function(df, group_var, ana_var){
df |>
group_by(pick({{ group_var }})) |>
summarise(
mean = mean({{ ana_var }}, na.rm=TRUE),
max = max({{ ana_var }}, na.rm=TRUE))
}
group_summ_3(d.class, c(sex, age), height)select, across等函数在指定变量时,
使用整洁选择方式。
如果变量是用整洁选择函数或者原样变量名方式输入的,
仍可以用{{ var_names }}这种格式。
例如, 下面的函数输入一个数据框, 输入一个分组用的变量名, 输入要计算汇总统计量的数值型变量, 输出分组汇总结果的数据框:
group_summ_4 <- function(df, group_var, ana_vars){
df |>
group_by({{ group_var }}) |>
summarise(across(
{{ ana_vars }},
list(avg = ~ mean(., na.rm=TRUE),
max = ~ max(., na.rm=TRUE))))
}
group_summ_4(d.class, sex, c(height, weight))## # A tibble: 2 × 5
## sex height_avg height_max weight_avg weight_max
## <fct> <dbl> <dbl> <dbl> <dbl>
## 1 M 63.9 72 109. 150
## 2 F 60.6 66.5 90.1 112.其中的group_by是基于数据掩码的,
across是基于整洁选择的。
在across中如果输入字符型变量,
则不能用{{ var_names }}这种方法,
而是用all_of(var_names),
如:
group_summ_5 <- function(df, group_var, ana_vars){
df |>
group_by({{ group_var }}) |>
summarise(across(
all_of(ana_vars),
list(avg = ~ mean(., na.rm=TRUE),
max = ~ max(., na.rm=TRUE))))
}
ana_vars <- c("height", "weight")
group_summ_5(d.class, sex, ana_vars)## # A tibble: 2 × 5
## sex height_avg height_max weight_avg weight_max
## <fct> <dbl> <dbl> <dbl> <dbl>
## 1 M 63.9 72 109. 150
## 2 F 60.6 66.5 90.1 112.在自定义函数中使用dplyr功能, 可参考Programming with dplyr。 在自定义函数中使用tidyr功能, 可参考programming with tidyr。
24.6 数据分析流程控制
一个完整的数据分析过程, 经常需要反复地修改程序, 更新数据。 如果每次都重新运行整个的数据整理、概括、建模、结果呈现的代码, 会花费很多时间, 中间某一步骤出错时如果修改程序重新运行, 就使得前面步骤白作, 如果人为控制重新运行哪些代码, 容易犯错使得利用了旧的结果。
解决这样的问题的工具是make类的软件, 这些软件规定各个任务之间的依赖关系, 当任务依赖的数据和代码更新时自动管理需要重新运行哪些程序。 R提供了targets包可以实现这样的任务管理功能, 规定了数据、代码、结果的依赖关系后可以仅运行受到影响的部分。
24.7 基本R的汇总功能
24.7.1 summary()函数
对数值型向量x,用summary(x)可以获得变量的平均值、中位数、
最小值、最大值、四分之一和四分之三分位数。 如
## Min. 1st Qu. Median Mean 3rd Qu. Max.
## 12.58 43.77 93.40 110.08 157.18 330.24## Min. 1st Qu. Median Mean 3rd Qu. Max.
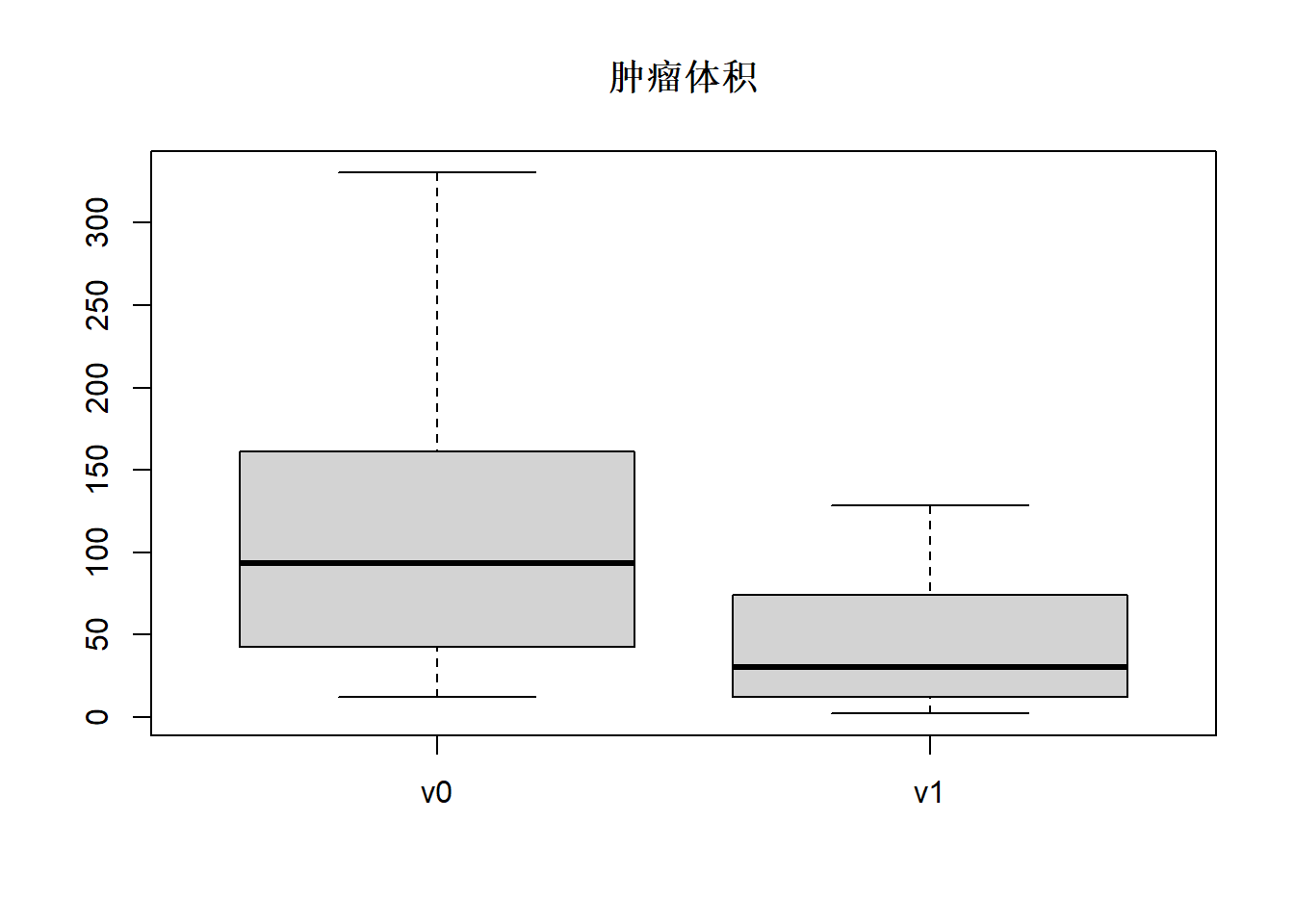
## 2.30 12.73 30.62 44.69 72.94 128.34v0是放疗前的肿瘤体积, v1是放疗后的体积, 可以看出放疗后体积减小了很多。
可以用盒形图表现类似的信息,如
对一个数据框d, 用summary(d)可以获得每个连续型变量的基本统计量,
和每个离散取值变量的频率。如
## id age sex type
## Min. : 1.00 Min. :49.00 Length:34 Length:34
## 1st Qu.: 9.25 1st Qu.:55.00 Class :character Class :character
## Median :17.50 Median :67.00 Mode :character Mode :character
## Mean :17.50 Mean :64.13
## 3rd Qu.:25.75 3rd Qu.:70.00
## Max. :34.00 Max. :79.00
## NA's :11
## v0 v1
## Min. : 12.58 Min. : 2.30
## 1st Qu.: 43.77 1st Qu.: 12.73
## Median : 93.40 Median : 30.62
## Mean :110.08 Mean : 44.69
## 3rd Qu.:157.18 3rd Qu.: 72.94
## Max. :330.24 Max. :128.34
## 对数据框d,用str(d)可以获得各个变量的类型和取值样例。 如
## spec_tbl_df [34 × 6] (S3: spec_tbl_df/tbl_df/tbl/data.frame)
## $ id : num [1:34] 1 2 3 4 5 6 7 8 9 10 ...
## $ age : num [1:34] 70 70 69 68 67 75 52 71 68 79 ...
## $ sex : chr [1:34] "F" "F" "F" "M" ...
## $ type: chr [1:34] "腺癌" "腺癌" "腺癌" "腺癌" ...
## $ v0 : num [1:34] 26.5 135.5 209.7 61 237.8 ...
## $ v1 : num [1:34] 2.91 35.08 74.44 34.97 128.34 ...
## - attr(*, "spec")=
## .. cols(
## .. id = col_double(),
## .. age = col_double(),
## .. sex = col_character(),
## .. type = col_character(),
## .. v0 = col_double(),
## .. v1 = col_double()
## .. )
## - attr(*, "problems")=<externalptr>用head(d)可以返回数据框(或向量、矩阵)的前几行,
用tail(d)可以返回数据框的后几行。
24.7.2 连续型变量概括函数
对连续取值的变量x,
可以用mean, std, var, sum, prod,
min, max等函数获取基本统计量。
加na.rm=TRUE选项可以仅对非缺失值计算。
sort(x)返回排序后的结果。
rev(x)把x所有元素次序颠倒后返回。
quantile(x, c(0.05, 0.95))可以求x的样本分位数。
rank(x)对x求秩得分(即名次,但从最小到最大排列)。
24.7.3 分类变量概括
分类变量一般输入为因子。
对因子或其它向量x,
table(x)返回x的每个不同值的频率(出现次数),
结果为一个类(class)为table的一维数组。
每个元素有对应的元素名,为x的各水平值。
如
##
## F M
## 13 21## F
## 13对单个分类变量, table结果是一个有元素名的向量。
用as.data.frame()函数把table的结果转为数据框:
## Var1 Freq
## 1 F 13
## 2 M 21用prop.table()将频数转换成百分比:
##
## F M
## 0.3823529 0.6176471table作的单变量频数表可以用barplot表现为图形,如:
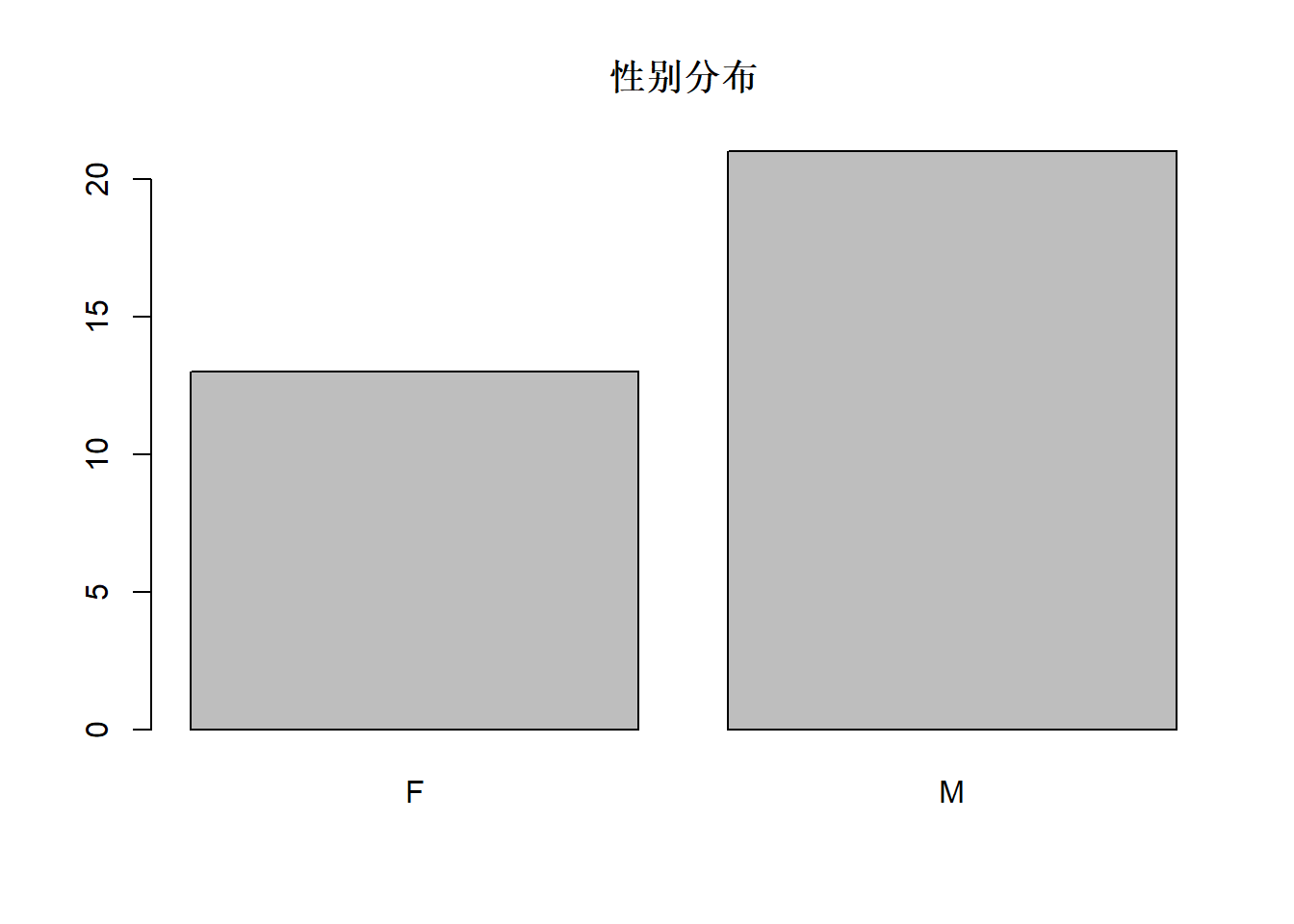
对两个分类变量x1和x2,
其每个组合的出现次数可以用table(x1,x2)函数统计, 结果叫做列联表。
如
## type
## sex 鳞癌 腺癌
## F 4 9
## M 18 3结果是一个类为table的二维数组(矩阵),
每行以第一个变量x1的各水平值为行名,
每列以第二个变量x2的各水平值为列名。
这里用了with()函数引入一个数据框,
后续的参数中的表达式可以直接使用数据框的变量。
对两个分类变量, table结果是一个矩阵。
用as.data.frame函数把table的结果转为数据框:
## sex type Freq
## 1 F 鳞癌 4
## 2 M 鳞癌 18
## 3 F 腺癌 9
## 4 M 腺癌 3列联表的结果可以用条形图表示。如
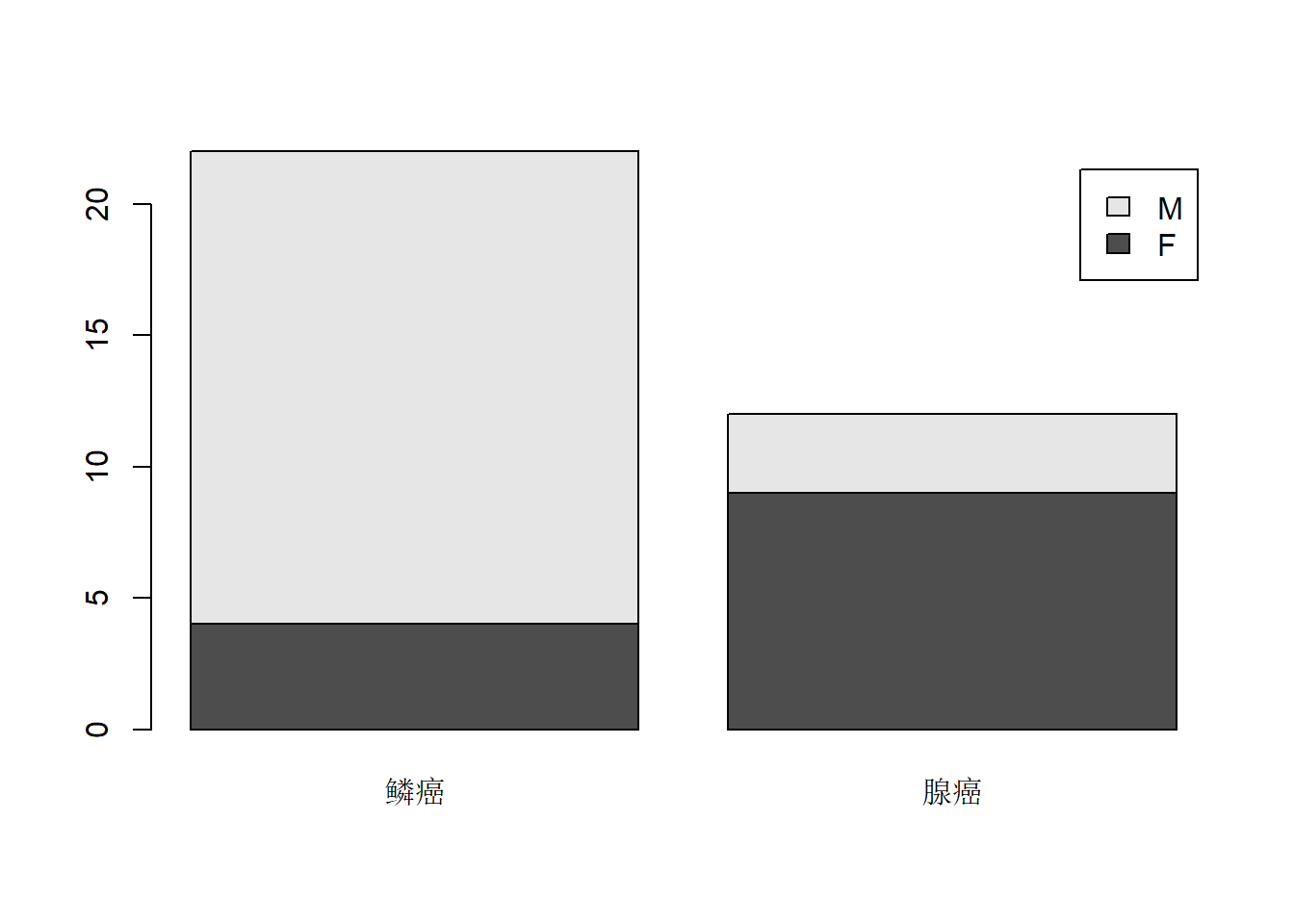
或
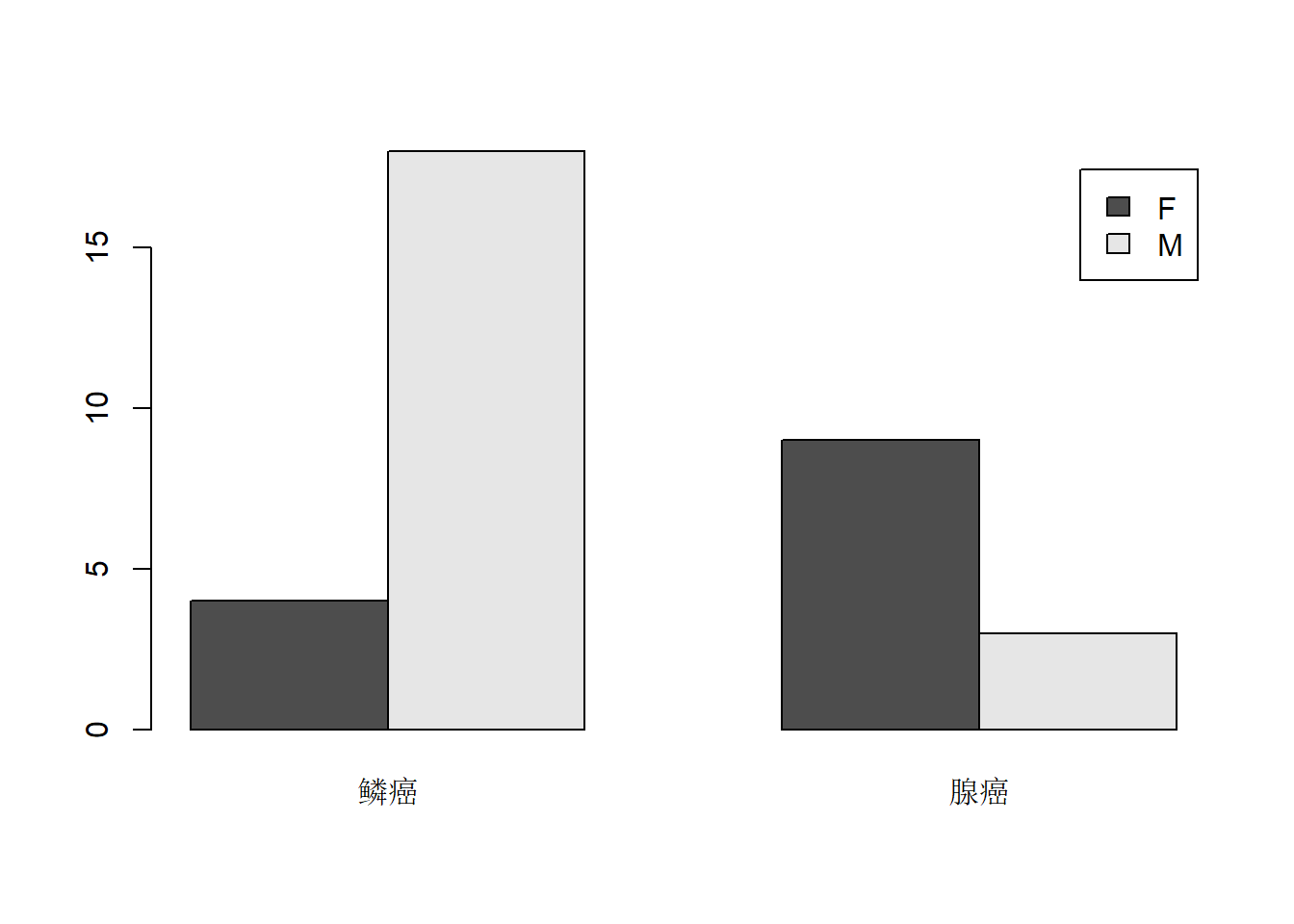
对于table()的结果列联表,
可以用addmargins()函数增加行和与列和。 如
## type
## sex 鳞癌 腺癌 Sum
## F 4 9 13
## M 18 3 21
## Sum 22 12 34用margin.table()可以计算列联表行或列的和并返回,如
## sex
## F M
## 13 21## type
## 鳞癌 腺癌
## 22 12用prop.table(r)把一个列联表r转换成百分比表。 如
## type
## sex 鳞癌 腺癌
## F 0.11764706 0.26470588
## M 0.52941176 0.08823529用prop.table(res,1)把列联表res转换成行百分比表。
用prop.table(res,2)把列联表res转换成列百分比表。 如
## type
## sex 鳞癌 腺癌
## F 0.3076923 0.6923077
## M 0.8571429 0.1428571## type
## sex 鳞癌 腺癌
## F 0.1818182 0.7500000
## M 0.8181818 0.2500000在有多个分类变量时,
用as.data.frame(table(x1, x2, x3))
形成多个分类变量交叉分类的频数统计数据框。
dplyr包的count()功能与table()类似。
24.7.4 数据框概括
用colMeans()对数据框或矩阵的每列计算均值,
用colSums()对数据框或矩阵的每列计算总和。
用rowMeans()和rowSums()对矩阵的每行计算均值或总和。
数据框与矩阵有区别,
某些适用于矩阵的计算对数据框不适用,
例如矩阵乘法。
用as.matrix()把数据框的数值子集转换成矩阵。
对矩阵,用apply(x, 1, FUN)对矩阵x的每一行使用函数FUN计算结果,
用apply(x, 2, FUN)对矩阵x的每一列使用函数FUN计算结果。
如果apply(x,1,FUN)中的FUN对每个行变量得到多个\(m\)结果,
结果将是一个矩阵,行数为\(m\),列数等于nrow(x)。
如果apply(x,2,FUN)中的FUN对每个列变量得到多个\(m\)结果,
结果将是一个矩阵,行数为\(m\),列数等于ncol(x)。
例如:
apply(as.matrix(iris[,1:4]), 2,
function(x)
c(n=sum(!is.na(x)),
mean=mean(x, na.rm=TRUE),
sd=sd(x, na.rm=TRUE)))## Sepal.Length Sepal.Width Petal.Length Petal.Width
## n 150.0000000 150.0000000 150.000000 150.0000000
## mean 5.8433333 3.0573333 3.758000 1.1993333
## sd 0.8280661 0.4358663 1.765298 0.762237724.8 用基本R作分类汇总
24.8.1 用tapply()分组汇总
用tapply()函数进行分组概括, 格式为:
其中X是一个向量, INDEX是一个分类变量, FUN是概括统计函数。
比如,下面的程序分性别组计算疗前体积的均值:
## F M
## 113.2354 108.121424.8.2 用aggregate()分组概括数据框
aggregate函数对输入的数据框用指定的分组变量(或交叉分组)
分组进行概括统计。
例如,下面的程序按性别分组计算年龄、疗前体积、疗后体积的平均值:
## sex age v0 v1
## 1 F 66.14286 113.2354 42.65538
## 2 M 63.25000 108.1214 45.95524aggregate()第一个参数是数据框,
第二个参数是列表,列表元素是用来分组或交叉分组的变量,
第三个参数是概括用的函数,
概括用的函数的选项可以在后面给出。
可以交叉分组后概括,如
## sex type v0 v1
## 1 F 鳞癌 126.99250 45.54750
## 2 M 鳞癌 113.55722 49.65556
## 3 F 腺癌 107.12111 41.37000
## 4 M 腺癌 75.50667 23.7533324.9 用plyr包进行分类概括
plyr是一个专注于分组后分别分析然后将分析结果尽可能合理地合并的扩展包, 功能强大, dplyr包仅针对数据框,使用更方便,但是对于复杂情况功能不如plyr包强。 plyr包已经被dplyr、purrr包代替,不再继续开发新版本。 这部分内容仅作为备忘, 读者可以跳过。
plyr的输入支持数组、数据框、列表, 输出支持数组、数据框、列表或无输出。 分组分析的函数输出格式需要与指定的输出格式一致。
这里主要介绍从数据框分组概括并将结果保存为数据框的方法,
使用plyr包的ddply()函数。
实际上,dplyr包的这种功能更方便。
plyr包的优点是可以自定义概括函数,
使得结果表格符合用户的预期,
处理多个变量时程序更简洁。
plyr包与dplyr包的函数名冲突比较大, 所以需要先卸载dplyr包再调用plyr包:
ddply()函数第一自变量是要分组的数据框,
第二自变量是分组用的变量名,
第三自变量是一个概括函数,
此概括函数以一个数据框子集(数据类型是数据框)为输入,
输出是一个数值、一个数值型向量或者一个数据框,
但最好是数据框。
例如,按性别分组,计算v0的平均值:
下面的程序按性别分组, 分别计算v0与v1的平均值:
下面的程序按性别分组,计算v0和v1的平均值、标准差:
f1 <- function(dsub){
tab <- tibble(
"变量"=c("v0", "v1"),
"均值"=c(mean(dsub[,"v0"], na.rm=TRUE),
mean(dsub[,"v1"], na.rm=TRUE)),
"标准差"=c(sd(dsub[,"v0"], na.rm=TRUE),
sd(dsub[,"v1"], na.rm=TRUE)))
tab
}
ddply(d.cancer, "sex", f1)注意f1()结果是一个数据框。
程序有些重复内容,对每个变量和每个统计量都需要分别写出,
如果这样用plyr包就不如直接用dplyr::summarise()了。
下面用vapply()简化程序。
按性别分组,然后v0、v1各自一行结果, 计算非缺失值个数、均值、标准差、中位数:
f2 <- function(d, variables){
d1 <- d[,variables,drop=FALSE]
nnotmiss <- vapply(d1, function(x) sum(!is.na(x)), 1L)
xm <- vapply(d1, mean, 0.0, na.rm=TRUE)
xsd <- vapply(d1, sd, 1.0, na.rm=TRUE)
xmed <- vapply(d1, median, 0.0, na.rm=TRUE)
data.frame(variable=variables,
n=nnotmiss,
mean=xm,
sd=xsd,
median=xmed)
}
ddply(d.cancer, "sex", f2, variables = c("v0", "v1"))f2()函数针对分组后的数据框子集,
这样的函数可以先在一个子集上试验。
在f2()函数中,
设输入的数据子集为d,
要分析的变量组成的数据框为d1,
用vapply()函数对d1的每一列计算一种统计量,
然后将每种统计量作为结果数据框的一列。
vapply()函数类似于lapply()和sapply(),
但是用第三个自变量表示要应用的变换函数的返回值类型和个数,
用举例的方法给出。
ddply()也可以对交叉分类后每个类分别汇总,
例如按照性别与病理类型交叉分组后汇总v0、v1:
上面的程序写法适用于已知要分析的变量名的情况。 如果想对每个数值型变量都分析, 而且想把要计算的统计量用统一的格式调用,可以写成:
f3 <- function(d){
ff <- function(x){
c(n=sum(!is.na(x)),
each(mean, sd, median)(x, na.rm=TRUE))
}
ldply(Filter(is.numeric, d), ff)
}
ddply(d.cancer, "sex", f3)ff()函数对输入的一个数值型向量计算4种统计量,
返回一个长度为4的数值型向量,
用来对分组后的数据子集中的一列计算4种统计量。
plyr包的each()函数接受多个函数,
返回一个函数可以同时得到这几个函数的结果,
结果中各元素用输入的函数名命名。
f3()函数中的Filter函数用于从列表或数据框中取出满足条件的项,
这里取出输入的数据子集d中所有的数值型列。
f3()函数中的ldply()函数接受一个列表或看成列表的一个数据框,
对数据框的每列应用ff()函数计算4种统计量,
然后合并所有各列的统计量为一个数据框,
结果数据框的每行对应于d中的一列。
程序中的ddply()函数接受一个数据框,
第二自变量指定用来将数据框分组的变量,
第三自变量f3()是对分组后的数据框子集进行分析的函数,
此函数接受一个数据框,输出一个数据框。
上面的程序也可以利用无名函数写成:
24.10 练习
把
patients.csv读入“d.patients”中, 并计算发病年龄、发病年、发病月、 发病年月(格式如“200702”表示2007年2月份)。把“现住地址国标”作为字符型,去掉最后两位,仅保留前6位数字, 保存到变量“地址编码”中。
按照地址编码和发病年月交叉分类汇总发病人数, 保存到数据框d.pas1中, 然后保存为CSV文件“分区分年月统计.csv”中。 要求结果有三列:“地址编码”、“发病年月”、“发病人数”。
按照地址编码和发病月分类汇总发病人数, 保存到数据框d.pas2中, 然后保存为CSV文件“分区分月统计.csv”中。 要求每个地址编码占一行, 各列为地址编码以及1、2、…………、12各月份, 每行为同一地址编码各月份的发病数。
按发病年月和性别汇总发病人数, 并计算同年月不分性别的发病总人数。 结果保存到数据框d.pas3中, 然后保存到CSV文件“分年月分性别统计.csv”中。 要求每个不同年月占一行, 变量包括年月、男性发病数、女性发病数、总计。
分析病人的职业分布,保存到数据框d.pas4中, 然后保存到CSV文件“职业构成.csv”中。 要求各列为职业、发病人数、百分比(结果乘以100并保留一位小数)。
把年龄分成0—9, 11—19, ……, 70以上各段, 保存为“年龄段”变量。 用年龄段和性别交叉汇总发病人数和百分比(结果乘以100并保留一位小数), 保存到“年龄性别分布.csv”中。 要求将每个年龄段的男性发病人数、发病率、女性发病人数、发病率存为一行。